「ADPリボシル化因子」の版間の差分
細 →細胞内局在 |
細編集の要約なし |
||
| (同じ利用者による、間の1版が非表示) | |||
| 8行目: | 8行目: | ||
英:ADP ribosylation factor<br> | 英:ADP ribosylation factor<br> | ||
英略語:Arf | 英略語:Arf | ||
{{box|text= ADPリボシル化因子(Arf)は、Rasスーパーファミリーに属する低分子量GTP結合タンパク質で、哺乳類ではArf1からArf6の6種類の分子(ヒトではARF2以外の5分子) | {{box|text= ADPリボシル化因子(Arf)は、Rasスーパーファミリーに属する低分子量GTP結合タンパク質で、哺乳類ではArf1からArf6の6種類の分子(ヒトではARF2以外の5分子)が存在する。ArfはGTP結合型とGDP結合型の2つのコンフォメーションを変換させて、細胞内小胞輸送やアクチン細胞骨格の再構築を制御する分子スイッチとして機能する。神経系において、Arfは軸索、樹状突起、棘突起の形成と維持、細胞移動、シナプス小胞サイクル、シナプス可塑性などの多彩な機能に関与する。「ADPリボシル化因子」の名称は、試験管内でのコレラ毒素の[[補酵素]]としての性質に由来するもので、必ずしも生理的な機能を反映するものではない。}} | ||
== 発見の経緯とその後 == | == 発見の経緯とその後 == | ||
| 26行目: | 26行目: | ||
== 細胞内局在 == | == 細胞内局在 == | ||
Arfは様々な組織に幅広く発現する<ref name=Hosaka1996><pubmed>8947846</pubmed></ref>。内因性Arfの細胞内局在は、分子間の高い同一性から特異的な[[抗体]]の作製が難しく、依然明らかにされていない。[[培養細胞]]への[[強制発現系]]を用いた局在解析から、Arf1-5は[[小胞体]]、[[ゴルジ装置]]、[[エンドソーム]]に主に局在するのに対して、Arf6は細胞膜とエンドソームに局在することが報告されている<ref name=D'Souza-Schorey2006><pubmed>16633337</pubmed></ref><ref name=Radhakrishna1997><pubmed>9314528</pubmed></ref>。[[CRISPR-Cas9]]を用いた[[ゲノム編集]]技術によりクラスI Arf (Arf1, Arf3)とクラスII Arf (Arf4, Arf5)のC末領域にタグをノックインした内因性Arfタンパク質の細胞内局在に関する[[超解像度顕微鏡]]解析の報告によると、[[HeLa細胞]]において、クラスIとクラスII Arfとも[[小胞体–ゴルジ体中間区画]]([[ERGIC]])、ゴルジ装置、[[管状小胞状]]の構造に部分的に共存しながら分布するが、同じ細胞小器官において異なるナノドメインへの局在も示すことから、クラスIとII のArfの各分子は、重複する機能とともに特異的な機能を持つものと考えられる<ref name=Wong-Dilworth2023><pubmed>37102998</pubmed></ref>。 | Arfは様々な組織に幅広く発現する<ref name=Hosaka1996><pubmed>8947846</pubmed></ref>。内因性Arfの細胞内局在は、分子間の高い同一性から特異的な[[抗体]]の作製が難しく、依然明らかにされていない。[[培養細胞]]への[[強制発現系]]を用いた局在解析から、Arf1-5は[[小胞体]]、[[ゴルジ装置]]、[[エンドソーム]]に主に局在するのに対して、Arf6は細胞膜とエンドソームに局在することが報告されている<ref name=D'Souza-Schorey2006><pubmed>16633337</pubmed></ref><ref name=Radhakrishna1997><pubmed>9314528</pubmed></ref>。[[CRISPR-Cas9]]を用いた[[ゲノム編集]]技術によりクラスI Arf (Arf1, Arf3)とクラスII Arf (Arf4, Arf5)のC末領域にタグをノックインした内因性Arfタンパク質の細胞内局在に関する[[超解像度顕微鏡]]解析の報告によると、[[HeLa細胞]]において、クラスIとクラスII Arfとも[[小胞体–ゴルジ体中間区画]]([[ERGIC]])、ゴルジ装置、[[管状小胞状]]の構造に部分的に共存しながら分布するが、同じ細胞小器官において異なるナノドメインへの局在も示すことから、クラスIとII のArfの各分子は、重複する機能とともに特異的な機能を持つものと考えられる<ref name=Wong-Dilworth2023><pubmed>37102998</pubmed></ref>。 | ||
[[ファイル:Sakagami Arf Fig.png|サムネイル|'''図. マウスArfの活性化制御分子(GEFとGAP)ファミリーとドメイン構造'''<br>哺乳類のArfGEFは15分子6ファミリーからなり、GEF活性に必須の約200アミノ酸からなるSec7領域を持つ。ArfGAPは31分子10ファミリーに分類されている。<br>ALPS, amphipathic lipid packaging sensor; ANK, ankyrin repeats; BAR, Bin/Amphiphysin/Rvs domain; CC, coiled-coil motif; DCB, dimerization and cyclophilin-binding domain; FAT, focal adhesion targeting domain; HDS, homology downstream of Sec7; HUS, homology upstream of Sec7; IQ, IQ motif; PH, pleckstrin homology domain; RA, Ras-associating domain; SAM, sterile α motif domain; SH3, Src-homology 3 domain; SLD, synaptic localization domain | [[ファイル:Sakagami Arf Fig.png|サムネイル|'''図. マウスArfの活性化制御分子(GEFとGAP)ファミリーとドメイン構造'''<br>哺乳類のArfGEFは15分子6ファミリーからなり、GEF活性に必須の約200アミノ酸からなるSec7領域を持つ。ArfGAPは31分子10ファミリーに分類されている。<br>ALPS, amphipathic lipid packaging sensor; ANK, ankyrin repeats; BAR, Bin/Amphiphysin/Rvs domain; CC, coiled-coil motif; DCB, dimerization and cyclophilin-binding domain; FAT, focal adhesion targeting domain; HDS, homology downstream of Sec7; HUS, homology upstream of Sec7; IQ, IQ motif; PH, pleckstrin homology domain; RA, Ras-associating domain; SAM, sterile α motif domain; SH3, Src-homology 3 domain; SLD, synaptic localization domain]] | ||
== 活性化制御 == | == 活性化制御 == | ||
| 113行目: | 113行目: | ||
== 神経疾患との関わり == | == 神経疾患との関わり == | ||
=== 脳形成障害 === | === 脳形成障害 === | ||
Sheenら(2004)は、小脳症と脳室周囲異所性灰白質を持つトルコ家系からクラスI Arfに対するGEFであるBIG2/ARFGEF2のホモ接合型[[ミスセンス変異]]を同定した<ref name=Sheen2004><pubmed>14647276</pubmed></ref>。さらにゲノムの翻訳部領域においてミスセンス変異が生じることが通常ないゲノム領域(missense-depleted region)に着目した、先天性脳構造異常の患者に対する[[エクソーム解析]]により、[[MRI]]で脳室周囲結節性異所性灰白質を示す発達障害とともに[[ | Sheenら(2004)は、小脳症と脳室周囲異所性灰白質を持つトルコ家系からクラスI Arfに対するGEFであるBIG2/ARFGEF2のホモ接合型[[ミスセンス変異]]を同定した<ref name=Sheen2004><pubmed>14647276</pubmed></ref>。さらにゲノムの翻訳部領域においてミスセンス変異が生じることが通常ないゲノム領域(missense-depleted region)に着目した、先天性脳構造異常の患者に対する[[エクソーム解析]]により、[[MRI]]で脳室周囲結節性異所性灰白質を示す発達障害とともに[[注意欠如・多動性障害]]を示す男児からARF1のGDP結合部位のミスセンス変異(p.Tyr35His)が同定された<ref name=Ge2016><pubmed>28868155</pubmed></ref>。その後、ARF1遺伝子のde novoミスセンス変異がさらに13ヶ所見出されている<ref name=deSainteAgathe2023><pubmed>37185208</pubmed></ref><ref name=Ishida2023><pubmed>36345169</pubmed></ref>。ARF1遺伝子変異を持つ患者は、[[知的障害]]とともに小脳症、異所性灰白質、脳梁の菲薄化などの種々の程度の脳構造異常を伴う。小脳症と脳室周囲異所性灰白質の機序として、BIG2→ARF1経路の障害による脳室上皮細胞間の細胞接着を介した脳室帯構造維持の破綻や神経幹細胞の細胞増殖の障害とともに脳室帯離脱後の細胞移動障害などが考えられている。 | ||
また、[[常染色体顕性遺伝]]性の先天性脳形成異常の患者からARF3遺伝子の2種類のde novoのミスセンス変異が同定されている<ref name=Sakamoto2021><pubmed>34346499</pubmed></ref>。ARF3遺伝子変異患者が呈した脳形成障害は、進行性の[[大脳]]や[[脳幹]]の萎縮あるいは非進行性の小脳低形成で2症例間で異なるが、いずれの患者も著明な発育遅延、てんかん、知的障害を伴う。 | また、[[常染色体顕性遺伝]]性の先天性脳形成異常の患者からARF3遺伝子の2種類のde novoのミスセンス変異が同定されている<ref name=Sakamoto2021><pubmed>34346499</pubmed></ref>。ARF3遺伝子変異患者が呈した脳形成障害は、進行性の[[大脳]]や[[脳幹]]の萎縮あるいは非進行性の小脳低形成で2症例間で異なるが、いずれの患者も著明な発育遅延、てんかん、知的障害を伴う。 | ||
2025年6月10日 (火) 20:25時点における最新版
阪上 洋行
北里大学・医学部・解剖学
DOI:10.14931/bsd.11017 原稿受付日:2025年4月24日 原稿完成日:2025年4月27日
担当編集委員:和田 圭司(国立研究開発法人国立精神・神経医療研究センター)
英:ADP ribosylation factor
英略語:Arf
ADPリボシル化因子(Arf)は、Rasスーパーファミリーに属する低分子量GTP結合タンパク質で、哺乳類ではArf1からArf6の6種類の分子(ヒトではARF2以外の5分子)が存在する。ArfはGTP結合型とGDP結合型の2つのコンフォメーションを変換させて、細胞内小胞輸送やアクチン細胞骨格の再構築を制御する分子スイッチとして機能する。神経系において、Arfは軸索、樹状突起、棘突起の形成と維持、細胞移動、シナプス小胞サイクル、シナプス可塑性などの多彩な機能に関与する。「ADPリボシル化因子」の名称は、試験管内でのコレラ毒素の補酵素としての性質に由来するもので、必ずしも生理的な機能を反映するものではない。
発見の経緯とその後
ADPリボシル化因子(Arf)は、コレラ毒素Aサブユニットによるヘテロ三量体Gタンパク質GαsのADP-リボシル化に必要な補因子として、ウサギ肝臓とウシ脳から精製された[1][2][3]。精製されたArf自体がGTP結合能を持ち[4]、さらにSewellとKahn (1988年) [5]によりクローニングされたウシArf1の一次構造から、Rasや三量体Gタンパク質Gαsと相同性の高い配列を持つ低分子量GTP結合タンパク質であることが明らかになった。その後、哺乳類ではArf1-Arf6の6種(ヒトでは5種)のパラログの存在が明らかになっている[6]。
「ADPリボシル化因子」の名称は、試験管内でのコレラ毒素の補酵素としての性質に由来するもので、生理的な機能を反映するものではない。細胞内において、Arfは小胞輸送やアクチン細胞骨格の制御に関わる低分子量GTP結合タンパク質として機能している。コレラ毒素のADPリボシル化の補酵素としての性質に基づいたArfの本来の定義に該当する分子はArf1-Arf6の6分子(ヒトでは5分子)のみであるが、近年、Arf-like (Arl)、Arf-related protein 1 (ARFRP1)、secretion-associated and Ras-related protein 1 (SAR1)などの近縁分子が多数同定され、Arfスーパーファミリーとして広がりつつある。
ファミリー構成分子
哺乳類のArfファミリーは、異なる遺伝子から生じる6種のパラログ(Arf1-6)からなる[6]。ただし、ヒトARF2は偽遺伝子化しタンパク質として存在しない。6種のArf分子は一次配列とゲノム構造の類似性から3つのクラスに分類され、クラスIにはArf1、Arf2、Arf3、クラスIIにはArf4、Arf5、クラスIIIにはArf6が属する。クラスIは181アミノ酸、クラスII は180アミノ酸、唯一クラスIIIに属するArf6は175アミノ酸からなる。マウスArfにおいて同一クラス間で90-96%, クラスIとクラスII間では78-81%、クラスI/IIとクラスIII間では65-69%のアミノ酸の同一性がある。なお、記載法として、「ARF」はヒトの遺伝子あるいはタンパク質に対して使われ、「Arf」はヒト以外の種での名称、総称、活性などに対して用いられることが推奨されている[7]。
構造
Arfは、低分子量GTP結合タンパク質に共通の構造として、GTPおよびGDPとの結合により大きく立体構造を変化させてGTP依存的にエフェクターと相互作用するスイッチIとスイッチII領域を持つ[8][9]。さらに他の低分子量GTP結合タンパク質とは異なる特徴として、N末端の開始メチオニン残基が除去され、2番目のグリシン残基に14炭素鎖飽和脂肪酸のミリスチン酸(C14:0)が不可逆的にアミド結合で付加されている。さらにそれに続くN末端領域に両親媒性αヘリックス領域が存在し、Arfの脂質膜との相互作用とともにスイッチIとスイッチII領域の構造変化に関わる。脂質膜と相互作用するN末端領域とエフェクターと相互作用するスイッチ領域間の立体構造上の距離が短い(1-2nm)ため、他の低分子量GTP結合タンパク質に比べて、エフェクターは脂質膜のより近くにリクルートされ作用する。これらの性質はArfの下流エフェクターであるリン脂質代謝酵素やアダプタータンパク質によるリン脂質膜への作用を介した小胞輸送制御に密接に関連すると考えられている[8]。さらにGDP結合型とGTP結合型への変換に伴いスイッチIとスイッチII領域を繋ぐインタースイッチ領域も大きく立体構造を変化させるのもArfの構造上の特徴として挙げられる。
下流エフェクター
他の低分子量GTP結合タンパク質と同様に、GTP結合型Arfは活性型として、種々のエフェクターを細胞膜や細胞小器官の脂質膜マイクロドメインにリクルートし、小胞輸送経路やアクチン細胞骨格の再構築を促す[10][11]。多数のエフェクター分子が同定されており、それぞれArf分子に対する選択性を持つが、主要なエフェクターとして、①コートタンパク質(COPI複合体、Golgi-localized, γ-ear-containing, Arf-binding protein (GGA)、アダプタータンパク質複合体(AP-1, 2, 3, 4)、②リン脂質修飾酵素(ホスホリパーゼD、ホスファチジルイノシトール 4-リン酸5-キナーゼ (PIP5K)、ホスファチジルイノシトール 4-キナーゼ)、③モータータンパク質(MKLP1)とアダプター分子(FIP3/4, JIP3/4)、④小胞繋留因子 (エクソシスト複合体サブユニットSec10、ゴルジンGMAP210、GARP/EARP複合体サブユニットVps52)などが挙げられる。また、GDP型Arfに選択的に結合する分子も同定されており、例えばGDP結合型Arf6の場合、アダプタータンパク質Fe65[12]、Rac/Rho-GEFであるKalirin[13]、Rab-GAPであるTBC1D24[14]などが挙げられる。これらの事実から、古典的なGDP-GTPサイクルのドグマにおいて不活性型とされるGDP型Arfが、シグナル経路の調節に積極的に関与している可能性が示唆される。
細胞内局在
Arfは様々な組織に幅広く発現する[6]。内因性Arfの細胞内局在は、分子間の高い同一性から特異的な抗体の作製が難しく、依然明らかにされていない。培養細胞への強制発現系を用いた局在解析から、Arf1-5は小胞体、ゴルジ装置、エンドソームに主に局在するのに対して、Arf6は細胞膜とエンドソームに局在することが報告されている[10][15]。CRISPR-Cas9を用いたゲノム編集技術によりクラスI Arf (Arf1, Arf3)とクラスII Arf (Arf4, Arf5)のC末領域にタグをノックインした内因性Arfタンパク質の細胞内局在に関する超解像度顕微鏡解析の報告によると、HeLa細胞において、クラスIとクラスII Arfとも小胞体–ゴルジ体中間区画(ERGIC)、ゴルジ装置、管状小胞状の構造に部分的に共存しながら分布するが、同じ細胞小器官において異なるナノドメインへの局在も示すことから、クラスIとII のArfの各分子は、重複する機能とともに特異的な機能を持つものと考えられる[16]。
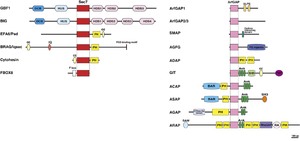
哺乳類のArfGEFは15分子6ファミリーからなり、GEF活性に必須の約200アミノ酸からなるSec7領域を持つ。ArfGAPは31分子10ファミリーに分類されている。
ALPS, amphipathic lipid packaging sensor; ANK, ankyrin repeats; BAR, Bin/Amphiphysin/Rvs domain; CC, coiled-coil motif; DCB, dimerization and cyclophilin-binding domain; FAT, focal adhesion targeting domain; HDS, homology downstream of Sec7; HUS, homology upstream of Sec7; IQ, IQ motif; PH, pleckstrin homology domain; RA, Ras-associating domain; SAM, sterile α motif domain; SH3, Src-homology 3 domain; SLD, synaptic localization domain
活性化制御
ArfのGDP-GTPサイクルはグアニンヌクレオチド交換因子(GEF)とGTPase活性化因子(GAP)により厳密に調節されている。
Arf-GEF
1996年に酵母からGea1が、哺乳類からサイトヘジン-2 (ARNO)とBIG1のcDNAが最初にクローニングされた[17][18]。その一次構造の比較から、GDPからGTPへの交換活性に必須である約200アミノ酸からなるSec7ドメインの存在が明らかになった。Sec7ドメインを持つタンパク質は、ヒトでは15種存在し、ドメイン構造の特徴から、GBF1ファミリー、BIGファミリー、サイトへジンファミリー、BRAG/IQSECファミリー、EFA6/PSDファミリー、F-box only protein 8 (FBXO8)に分類される(図)[19][8][20]。
FBXO8以外のSec7タンパク質は、Arf各分子に対する基質親和性は異なるもののArfに対するグアニンヌクレオチド交換活性を持つ[19][8][20]。一方、FBXO8はSec7とともにF-boxドメインを持ち、GEFとしては機能せず、Arf6の非分解性のユビキチン化を介した活性の制御に関わる[21]。Arf-GEFは、Sec7ドメイン以外にコイルドコイルモチーフ、プレクストリン相同ドメイン(PH domain)、PDZ結合モチーフ、IQモチーフなどファミリーごとに特徴的なドメイン構造を持ち、Arf-GEFの細胞内局在や活性が制御されている。例えば、BRAG1とBRAG2は、C末端のPDZ結合モチーフを介してPSD-95との結合し、海馬神経細胞の興奮性シナプスのシナプス後肥厚部に局在する[22][23]。また、サイトへジン-2はN末端のコイルドコイルモチーフを介してタマリンと結合し代謝型グルタミン酸受容体と複合体を形成し、興奮性シナプスのペリシナプスに局在する[24][25]。一方、BRAG3はゲフィリンやジストロフィンとの結合を介して抑制性シナプスのシナプス後膜に選択的に局在する[26][27]。
Arf-GAP
一方、精製されたArfにはGTPase活性が検出されないことより[4]、GAPの存在がGTPの加水分解に必須であることが発見当初より予想されていた[4]。1994年にRandazzoとKahnによりウシ脳抽出液にホスファチジルイノシトール 4,5-ビスリン酸 (PI(4,5)P2) 存在下で活性化するArf-GAPの存在が示された[28]。同年、MaklerらによりArf1に対するGAP活性を指標に約49kDaのArf-GAPがラット肝臓より精製され[29]。そのアミノ酸の部分配列よりcDNAが初めてクローニングされた[30]。その結果、C4タイプのジンクフィンガー構造(CX2CX16CX2CX4R)からなるGAP活性に必須のArfGAPドメインが見出された。
現在、ArfGAPドメインを共通に持つArf-GAP分子はヒトで31種同定されている [31]。ドメイン構造の類似性からArfGAP1ファミリー、ArfGAP2/3ファミリ―、SMAPファミリー、AGFGファミリー、ADAPファミリー、GITファミリー、ACAPファミリー、ASAPファミリー、AGAPファミリー、ARAPファミリーの10種類に分類される(図)[31]。
ArfGAPのドメイン構造はArf-GEFと比べてより多彩で、例えばARAP1はArf-GAPドメインとともにRho-GAPドメインやRas結合ドメイン[32]を、GIT1はRac-GEFであるPIXと結合するSpa2相同ドメインなどを持ち[33]、Arfのoffスイッチの制御分子であるとともに、Arf-GAP自体がGTP結合型Arfのエフェクターとして他のシグナル経路とのクロストークを担うものと考えられている。また、ArfGAPドメインはArf-GAP分子に必須のものではなく、ArfGAPドメインを持たないC9orf72やELMOD2がArf-GAP活性を示すことが報告されており[34][35][36][37]、今後さらにArf-GAPファミリーが広がる可能性がある。
また、RabやRhoファミリーと異なり、Arf活性制御にはGDP結合型の低分子量GTP結合タンパク質と結合しGDP解離を抑制するGDP解離阻害因子(GDI, GDP dissociation inhibitor)やGDP型低分子量GTP結合タンパク質からGDIを解離させるGDI置換因子(GDF, GDI displacement factor)は存在しない。
全身ノックアウトマウスの表現型
これまでArf1, Arf4, Arf5, Arf6の全身型ノックアウトマウスの表現型が報告されている。
Arf1
胎生致死。胎生3.5日齢 (E3.5)の胚盤胞までは野生型と外見上の差はなく成長するが、着床後まもないE5.5においてKOマウスの71.4%に変性がみられ、E12.5にはKOマウスは消失し存在しない。胎生致死の原因は不明であるが、少なくともKOマウスの胚盤胞の栄養外胚葉や内細胞塊は野生型と同様に成長することが培養系実験で示されている[38] 。
Arf4
胎生致死。E9.5のKOマウスは野生型と比較して著明な成長遅延を示し、E10.5には全てのKOマウスが致死に至る。KOマウスの臓性内胚葉細胞において、大型のリソソーム(頂端液胞)の減少やエンドサイトーシス受容体メガリンの頂端領域での分布の減少などが認められることより、臓性内胚葉細胞における母体―胚子間の物質交換の障害が胎生致死の機序として考えられている[39]。
Arf5
KOマウスはメンデル則に従って誕生し正常に成長する。異常な表現型は認められていない[40]。
Arf6
胎生致死。KOマウスは胎生中期の肝細胞索の形成障害による肝臓の発達不全を示し、胎生中期(E13.5)から後期にかけて致死に至る[41]。また、E12.5のKOマウスにおいて脊髄交連神経の正中線での軸索投射異常が認められる[42]。
神経系での機能
神経上皮細胞
脳室帯の神経幹細胞である神経上皮細胞の細胞間および細胞外マトリックスとの細胞接着の障害は、脳室帯の構造維持とともに脳構造の形成に重要であり、その障害は異所性灰白質や小頭症などの脳形成障害を引き起こす。
2004年Sheenらは異所性灰白質を伴う小頭症の原因遺伝子としてクラスI ArfのGEFであるヒトBIG2 (ARFGEF2)を同定した[43]。マウスBIG2は胎生期の脳室帯に豊富に発現し、Arf-GEF (BIG1、BIG2、GBF1)に対する阻害剤ブレフェルジンAの胎児期脳室帯への投与やBig2ノックアウトマウスにおいて、神経上皮細胞間のN-カドへリンを介した細胞接着が障害され、脳室帯の構造の破綻が生じる[44][45]。その後、ヒトARF1も異所性灰白質の原因遺伝子として報告され[46]、BIG2によるARF1の活性化が、神経上皮細胞間のN-カドへリン依存的な細胞接着の制御を介して脳室帯の構造維持に関与する可能性が示唆される。
また、胎生期のマウス脳室帯の神経上皮細胞において、EphB2-ephrin B1シグナルは、Arf6の活性化による頂端面でのインテグリンβ1のエンドサイトーシスを抑制し、インテグリンの表面発現を適切に維持することにより、脳室帯の構造維持、非対称分裂、脳室帯から分裂後の細胞離脱を制御することが報告されている[47]。
神経細胞移動
中枢神経系において神経幹細胞の非対称分裂により生じた幼若神経細胞は、脳室帯を離脱し、先導突起の伸長と細胞体や細胞核の牽引を繰り返し、最終的に機能する部位へと細胞移動する。この細胞移動には、牽引力となるアクチン細胞骨格の再構築とともに、小胞輸送経路による先導突起方向への細胞膜成分の輸送、細胞接着分子や誘引因子に対する受容体などの極性配置の調節が重要な役割を果たす。特にヒトARF1とBIG2が異所性灰白質の原因遺伝子であること[46][43]から、Arfの神経移動の重要性が示唆されていた。
大脳皮質層形成過程で、興奮性神経細胞は多極性移動、ロコモーション、ターミナルトランスロケーションからなる移動様式を取る。子宮内電気穿孔法を用いた研究から、Arf1、Arf4、Arf6の神経移動への機能関与が明らかになっている[48][49][50][51]。異所性灰白質と関連するヒトArf1変異遺伝子のマウス脳室帯細胞への導入により、神経細胞移動の障害による大脳皮質の層形成異常が生じる。このArf1変異遺伝子を発現する移動神経細胞はゴルジ装置の拡大を示すことより、Arf1のゴルジ装置での輸送経路の調節を介した神経細胞移動への関与が示唆されている[51]。一方、Arf4は多極性移動、多極性から双極性への形態変換、それに続くロコモーションに、Arf6は多極性移動とともに多極性から双極性への形態変換の段階に関わる[49][50]。移動神経細胞において、Arf4とArf6はそれぞれ逆行性輸送経路およびリサイクリング経路を介したN-カドへリンの小胞輸送の制御により放射状グリアとの細胞接着性を調節する可能性が示唆されている[49][50]。また、神経細胞移動におけるArf6の活性化制御としてArf6-GAPであるACAP3の関与が報告されている[52]が、Arf6-GEFは不明である。
軸索形成
Arf6は、PIP5Kの活性化を介して、成長円錐でのアクチン細胞骨格を制御し、軸索の分岐や伸長を抑制的に調節することが海馬初代培養細胞において報告されている[53]。同様に脊髄神経節初代培養細胞においてもArf6の活性化は軸索の伸長に抑制的に作用する[54]。
Arf6ノックアウトマウスは、胎生12.5日齢の脊髄において脊髄交連神経の正中線での軸索投射異常を示す[42]。その機序として、脊髄交連神経の成長円錐において、Arf6が軸索反発因子スリットの受容体Robo1や誘引因子Wnt5aの受容体Frizzledの適切なタイミングでの軸索膜上での発現を調節することにより、正中線通過後の交連線維のネトリンに対する反発活性の獲得や吻側方向での軸索のターニングや伸長に関与する可能性が報告されている[42][55]。なお、交連軸索投射異常を示すショウジョウバエ変異体スクリーニングにより、Arf6-GEFであるBRAGファミリー分子のオルソログであるSchizo / Lonerが同定されている。Schizoは正中部グリア細胞において膜結合型スリットのArf6依存的なエンドサイトーシスを介して軸索の正中線の交差に関与する[56]。
軸索輸送
Arf6は、脊髄神経節ニューロンの軸索やPC12細胞の突起において、インテグリンを含む輸送小胞の逆行性輸送に関与する[54]。さらにGTP結合型Arf6は、エフェクターのJIP3/JIP4と結合することにより、JIP3/JIP4のダイナクチン/ダイニン複合体との相互作用とダイニンのモーター活性を促進し、オートファゴソームの逆行性軸索輸送を制御することが海馬初代培養細胞で報告されている[57]。軸索でのArf6の活性制御機構として、中枢神経系細胞において、Arf6-GEFであるEFA6Aは、軸索初節に豊富に局在し、軸索起部においてArf6の活性化を促し、軸索輸送を調節している可能性が報告されている[58]。
一方、Arf4とArf5のダブルノックアウトマウス (Arf4/5 DKO)は、プロパノールやガバペンチン反応性の本態性振戦様の不随意運動を示す。さらに、Arf4/5 DKOマウスの小脳プルキンエ細胞において、電位依存性ナトリウムチャネルNav1.6の軸索起始部での局在が障害され、自発的な連続発火が低下することから、クラスII Arfが小胞輸送経路を介した軸索起始部でのイオンチャネルの発現を制御する可能性が示唆されている[40]。
樹状突起形成
Arf1とArf6は異なるシグナル制御経路を介して樹状突起の発達を制御する。Arf1はゴルジ装置においてBIG2により活性化され、RhoA-mDia経路を介して、頂上樹状突起の基部へのゴルジ装置の極性配置を制御し、頂上樹状突起の伸長・分岐に関与する[59]。一方、Arf6の活性化は、初代海馬培養細胞の樹状突起の分岐形成[53]や海馬スライス培養でのCA1錐体神経細胞の樹状突起の網状分子層への伸長を抑制する[60]。樹状突起形成におけるArf6活性の制御因子としては、GEFとしてサイトへジン-1 [60], サイトへジン-2 [61]、EFA6A [62]が、GAPとしてADAP1 (centaurin α1) [63]とACAP3[64]の関与が報告されている。Arf6による樹状突起形成制御機構として、Rac1を介した細胞骨格の制御[61]、JIP3やFIP3によるモータータンパク質の活性調節を介した小胞輸送の制御[65][66], GDP結合型Arf6との結合能を持つRab-GAPのTBC1D24を介した経路の関与[48]が報告されている。
スパインの形成・維持
スパイン(棘突起)は樹状突起に沿ったキノコ状の突起で、興奮性シナプスの主要な場である。スパインはアクチン細胞骨格に富み、神経入力依存性に形態を変化させることが知られており(構造的可塑性)、長期的な伝達効率の変化(シナプス可塑性)と密接に関連する。また、その形態異常と種々の神経発達障害との病態機序との関連性が指摘されている。これまでにArf1とArf6のスパインの形成・維持への機能関与が報告されている。
Arf1はPICK1との相互作用を介して、PICK1によるArp2/3依存的なアクチン重合の阻害を調節することでスパイン構造の制御に関わる[67]。GTP型Arf1はPICK1と結合しAMPA型グルタミン酸受容体(AMPAR)をシナプス後肥厚部(PSD)で複合体を形成している。NMDA型グルタミン酸受容体(NMDAR)依存的LTDの過程で、NMDA刺激によりGTP型Arf1がGIT1などのArf-GAPによりGDP型に変換される結果、PICK1はArf1との結合から解放され、それに代わりArp2/3と結合し、Arp2/3によるアクチン線維の重合を抑制することで、スパイン頭部の退縮が引き起こされる。
一方、Arf6 は、初代海馬培養細胞において、スパインの発達段階に応じて双方向性の作用を示す[68][69][70]。Arf6の活性化は、スパイン形成期(培養11-15日)においてフィロポディアからスパインへの形成に促進的な作用を示すのに対して、スパイン維持期(培養17-20日)にはスパインの維持に対して抑制的に作用する[68][69][70]。発達期におけるArf6の活性化によるスパイン形成・成熟の促進機序としては、ホスホリパーゼDの活性化を介したRac経路によるアクチン細胞骨格の再編成[69]や、細胞接着分子テレンセファリンの細胞膜からの取り込みによるフィロポディアからスパインへの成熟の促進[71]が考えられている。一方、Arf6による成熟期のスパイン維持の抑制機序として、成熟に伴うRacの発現低下とRhoの発現上昇によるRhoに対するArf6依存的なRac活性化経路の相対的な低下[69]、GDP型Arf6の結合タンパク質である家族性てんかん原因遺伝子産物TBC1D24の関与[72]が報告されている。Arf6のスパイン制御の上流活性化制御分子としては、Arf6-GEFとしてEFA6A[68][71]、BRAG2[73]が、Arf6-GAPとしてGIT1[74]、AGAP1[75]、ADAP1[76]が関与する。
神経終末部でのシナプス小胞サイクル
シナプス小胞サイクルは、シナプス小胞のシナプス前膜へのドッキングと融合、エンドサイトーシスによる膜成分の回収、再利用などの種々のステップからなり、神経伝達物質の持続的な放出を可能にするための神経細胞特有の小胞輸送経路である。
Asheryら[77]は、アフリカツメガエルの神経筋接合部において、脊髄運動ニューロンへのサイトへジン-1 / msec7-1の過剰発現により、骨格筋の自発性シナプス電流頻度の増加と運動ニューロン刺激により誘発される興奮性シナプス後電位(EPSC)の振幅が増加することから、Arfの活性化がシナプス前部での神経伝達物質の放出に関与する可能性を初めて報告した。Kraussら[78]は、精製したシナプトソーム膜を用いたクラスリン被覆小胞の再構築実験系で、GTP結合型Arf6がPIP5Kγの活性化を介してPI(4,5)P2の産生を増加させることにより、クラスリン被覆小胞の形成を促進することを示した。さらに、Tagliattiら[79]は、海馬初代培養細胞においてArf6のノックダウンにより、神経終末においてシナプス小胞の減少、エンドソーム様膜小器官の出現、アクティブゾーンにドッキングするシナプス小胞の増加が生じることが報告した。一連の研究から、Arf6はシナプス小胞のドッキングの調節やシナプス前膜からクラスリン依存的に回収されたシナプス小胞の膜成分の再利用のためのリサイクリング経路の決定などの種々のシナプス小胞サイクルの段階に関与するものと考えられている。サイトへジン阻害剤SecinH3によりArf6ノックダウンと同じ表現型を示すことから、Arf6の上流活性化因子としてサイトへジンファミリーが考えられる[79]。また、Arf-GAPであるGITファミリー分子は、アクティブゾーンの構成分子としてPiccoloと複合体を形成し[80]、ヘルドのカリックスシナプス前終末においてシナプス小胞の放出確率の調節に関与する[81]。
一方、Arf1のラット副腎髄質褐色細胞腫由来PC12細胞における有芯小胞の形成への機能関与の報告はある[82]が、クラスI/II Arfの神経細胞のシナプス小胞サイクルへの関与に関する実験証拠はない。
シナプス可塑性
シナプス可塑性のうち、長期増強 (LTP)と長期抑圧 (LTD)は、シナプス刺激に伴うシナプス後膜でのAMPARの長期的な発現量の増加と減少によりそれぞれ引き起こされるが、小胞輸送経路はAMPARのシナプス後膜での発現量を調節する主要な制御機構である。さらに、樹状突起スパイン内部のアクチン細胞骨格の再構築は、シナプス可塑性に伴う形態の変化とともに、細胞膜の湾曲、取り込み小胞の切断や輸送を介してシナプス後膜でのAMPARの動態と密接に関連する。
Arf1とArf6は、異なる機構を介して海馬の長期抑圧(LTD)に関与することが報告されている[67][83]。GTP結合型Arf1は、PICK1と結合しAMPARと複合体を形成しているが、NMDA刺激によりGIT1などによりGDP結合型に変換される結果、PICK1との結合が解除される。開放されたPICK1はArp2/3と結合しArp2/3依存的アクチン重合を阻害する。これによりスパインの退縮とともにAMPARのGluA2/3サブユニットのエンドサイトーシスが促進され、 NMDAR依存的な海馬LTDが生じる[67]。一方、Arf6-GEFであるBRAG2は代謝型グルタミン酸受容体 (mGluR)刺激によりAMPARと複合体を形成し、Arf6をGTP型に変換する。その結果、活性化したArf6はAMPARのクラスリン依存的なエンドサイトーシスを促進することにより、海馬シャーファー側枝-CA1シナプスでのmGluR依存的なLTDに関与する[83]。また、BRAG2-Arf6経路は、mGluR依存的LTDのみならず、NMDA依存的LTDにおけるAMPARのエンドサイトーシスにも関与する[83]。
一方、海馬LTPへのArf小胞輸送の機能関与は、ブレフェルジンAによるクラスI/II Arfの活性化の阻害[84]やArf6-GAPであるADAP1ノックアウトマウス[76]やAGAP3のノックダウン実験[85]から示唆されているが、直接的な実験証拠はない。
髄鞘形成
髄鞘は支持細胞(末梢神経ではシュワン細胞、中枢神経系ではオリゴデンドロサイト)の細胞膜からなる軸索を包む層状構造で、跳躍伝導や軸索の機械的な損傷から保護する役割を持つ。シュワン細胞の髄鞘形成には、Neuregulin-1とErb2/3を介する軸索―グリア間相互作用が不可欠である。山内らの一連の研究[86]から、シュワン細胞において、Arf6は、Fynによるリン酸化により活性化したサイトへジン-1とサイトへジン-2の作用でGTP結合型に変換され、アクチン細胞骨格の制御や髄鞘膜への膜成分の小胞輸送を介して髄鞘形成に促進的に作用する[87][88]。坐骨神経において、サイトへジン-1の発現は生後直後にピークを示し発達とともに徐々に低下するのに対して、サイトへジン-2の発現は生後の発達に伴い増加することから[89][88]、サイトへジン-1が髄鞘形成の早期に、サイトへジン-2が髄鞘形成の中後期にArf6の活性化因子として機能する。さらに、胎生12日齢のサイトへジン-1のノックアウトマウスにおいて、未熟なシュワン細胞の末梢神経に沿った腹側への移動の遅延が観察され、Fyn→サイトヘジン-1→Arf6経路が発生期の未熟なシュワン細胞の細胞移動にも関与することが示唆されている[88]。
一方、BIG1により活性化されたArf1は、ミエリンタンパク質ゼロ(MPZ)などの髄鞘タンパク質のアダプタータンパク質複合体AP-1依存的な小胞輸送を介して、髄鞘の初期形成段階に関わる[90]。
中枢神経系におけるオリゴデンドロサイトによる髄鞘形成にも、Arf6はシュワン細胞のそれとは異なる制御機構を介して機能関与する。未熟なオリゴデンドロサイトにおいてArf6は、ACAP2による不活性化状態からサイトへジン-2により活性化され、オリゴデンドロサイトの分化と髄鞘形成を促進的に作用する[91]。さらに、ネスチン-Creマウスとの交配による中枢神経系特異的Arf6ノックアウトマウスは、脳梁や海馬采での有髄神経の割合と髄鞘の厚さの低下を示す。この髄鞘形成障害はタウ-Creマウスとの交配による神経細胞特異的Arf6ノックアウトでも観察されるが、2',3'-環状ヌクレオチド3'-ホスホジエステラーゼ (CNP)-Creマウスとの交配によるオリゴデンドロサイト特異的ノックアウトでは観察されないことから、神経細胞のArf6によるオリゴデンドロサイトの髄鞘形成の調節機構の存在が考えられた。この分子機序として、神経細胞に発現するArf6が神経細胞からの線維芽細胞成長因子2 (FGF2)などの液性因子の分泌を調節することにより、オリゴデンドロサイト前駆細胞の細胞移動と髄鞘形成を間接的に制御する可能性が示されている[92]。
神経疾患との関わり
脳形成障害
Sheenら(2004)は、小脳症と脳室周囲異所性灰白質を持つトルコ家系からクラスI Arfに対するGEFであるBIG2/ARFGEF2のホモ接合型ミスセンス変異を同定した[43]。さらにゲノムの翻訳部領域においてミスセンス変異が生じることが通常ないゲノム領域(missense-depleted region)に着目した、先天性脳構造異常の患者に対するエクソーム解析により、MRIで脳室周囲結節性異所性灰白質を示す発達障害とともに注意欠如・多動性障害を示す男児からARF1のGDP結合部位のミスセンス変異(p.Tyr35His)が同定された[93]。その後、ARF1遺伝子のde novoミスセンス変異がさらに13ヶ所見出されている[94][46]。ARF1遺伝子変異を持つ患者は、知的障害とともに小脳症、異所性灰白質、脳梁の菲薄化などの種々の程度の脳構造異常を伴う。小脳症と脳室周囲異所性灰白質の機序として、BIG2→ARF1経路の障害による脳室上皮細胞間の細胞接着を介した脳室帯構造維持の破綻や神経幹細胞の細胞増殖の障害とともに脳室帯離脱後の細胞移動障害などが考えられている。
また、常染色体顕性遺伝性の先天性脳形成異常の患者からARF3遺伝子の2種類のde novoのミスセンス変異が同定されている[95]。ARF3遺伝子変異患者が呈した脳形成障害は、進行性の大脳や脳幹の萎縮あるいは非進行性の小脳低形成で2症例間で異なるが、いずれの患者も著明な発育遅延、てんかん、知的障害を伴う。
先天性知的障害
2008年に重度の知的障害と発育障害を伴うヒプスアリスミアの脳波所見を示す点頭てんかん(ウエスト症候群)様の女児において、X染色体と20番染色体間との転座(t(X;20)(p11.2;q11.2))が見出され、そのX染色体側の切断点にArf-GEFであるBRAG1/IQSEC2遺伝子が位置することが明らかになった[96]。さらに2009年にShoubridgeらは、X連鎖性非症候群性知的障害を持つ4家系の全X染色体エクソーム解析によりBRAG1/IQSEC2の4種のミスセンス変異を報告した[97]。4種のミスセンス変異のうち3カ所はグアニンヌクレオチド交換能に必須であるSec7ドメインに、残りの1カ所はカルモジュリン結合部位であるIQモチーフに存在し、いずれもArf6に対するGEF活性の低下を招く。現在、BRAG1/IQSEC2変異は70種類以上報告され、主要な先天性知的障害原因遺伝子のひとつとなっている。BRAG1/IQSEC2変異患者は、知的障害とともに、てんかんや自閉症スペクトラムを含む精神障害を伴うことが多い。またIQSEC2/BRAG1領域はX染色体不活化を逃れる領域で、BRAG1/IQSEC2のヘテロ接合性変異を持つ女性患者は様々な度合いの症状を示す[98]。さらにBRAG/IQSECファミリーに属するBRAG2/IQSEC1とBRAG3/IQSEC3も常染色体潜性遺伝形式を示す知的障害の原因遺伝子として同定されている[73][99]。
アルツハイマー病
アルツハイマー病の主要な病理学的特徴である老人斑の主成分であるアミロイドβタンパク質(Aβ)は、アミロイド前駆タンパク質(APP)がβセクレターゼ(BACE1)とγセクレターゼの2種類のタンパク質分解酵素により段階的に切断され産生される。APPとBACE1は、いずれも一回膜貫通型タンパク質で細胞膜とエンドソーム間を小胞輸送される過程で、同じ細胞内コンパートメントで出会いAPPの分解反応が起こる。
Arf6のAβ産生への関与に関して、Arf6がBACE1のクラスリン非依存的経路を介した細胞膜から細胞内への取り込みと初期エンドソームへの輸送を制御することにより、初期エンドソームでのAPPの切断を制御することがHeLa細胞を用いた発現系で初めて報告された [100]。しかしながら、その後、BACE1の細胞膜からの取り込みの主要な経路はクラスリン依存的経路であるとの報告がある[101]。また、BACE1はC末細胞内領域の酸性クラスター・ジロイシンモチーフ(DISLL)を介してArfの主要な下流エフェクターであるコートタンパク質GGAと相互作用することにより、ArfによるBACE1の小胞輸送の制御を介したAβ産生への関与の可能性も報告されている[102]。
一方、APPのArf6によるマクロピノサイトーシス経路を介した細胞内への取り込みが報告されている[103]。APPはアダプタータンパク質であるMintタンパク質(Mint1-3)やFe65との結合モチーフ(XYNPXY)をC末細胞内領域に持ち、これらのアダプタータンパク質との相互作用を介した細胞膜への小胞輸送の制御を受けることが報告されている[104][105]。興味深いことにMintとFe65はいずれもArfの結合タンパク質(MintはすべてクラスのGTP結合型Arfとの結合能を、Fe65はGDP結合型Arf6との結合能を持つ)であり、APPの細胞内輸送におけるArfの関与の可能性が示唆されている[12][106]。
また、Arf1とArf4が、新たに合成されたBACE1のゴルジ装置から細胞膜への輸送を介して、APPのプロセッシングに関与することがHeLa細胞で報告されている[107]。また、クラスIとII ArfのGEFであるGBF1がAPPのゴルジ装置から細胞膜への輸送に関与することが、大脳皮質初代培養系とSH-SY5Y細胞を用いた実験系で報告されている[108]。いずれも生体での機能的重要性は不明である。
さらに、アルツハイマー病患者の海馬CA3-CA4領域の錐体ニューロンにおけるArf6の免疫染色性の増加が報告されているが、症例数は少数(n=8)で信頼性に乏しい[103]。
筋萎縮性側索硬化症および前頭側頭型認知症
C9orf72遺伝子のイントロン1のGGGGCCの繰り返し配列の異常な伸長は、筋萎縮性側索硬化症と前頭側頭葉型認知症の最も多い遺伝的要因の一つである。C9orf72の生理機能に関しては不明な点が多いが、C9orf72との結合タンパク質の探索により、Arf1とArf6が同定されている[109]。また、脊髄運動ニューロンの初代培養系において、C9orf72はArf6の活性化を抑制することにより、Arf6の下流エフェクターであるRac1-LIMキナーゼによるコフィリンのリン酸化を抑制する。その結果として成長円錐でのアクチンのリモデリングと軸索の伸長が促進する[109]。さらにC9orf72はArfGAPドメインを持たないが、SMCR8およびWDR41と複合体を形成し、Arf1、Arf5、Arf6に対するArf-GAP活性を示す[36][37]。これらの結果に一致して、運動ニューロン細胞株NSC-34細胞においてC9orf72の発現抑制によりGTP結合型Arf6が増加すること、C9orf72遺伝子変異を持つALS患者由来iPS細胞における軸索伸長の抑制が恒常活性型Arf6の発現により回復することが示されている[109]。しかしながら、C9orf72遺伝子を原因とする筋萎縮性側索硬化症および前頭側頭葉型認知症の発症や病態におけるArfの役割は不明である。
α-シヌクレイン関連疾患
α-シヌクレインが細胞内に凝集し蓄積することにより引き起こされる細胞死は、パーキンソン病、レビー小体型認知症、多系統萎縮症などのシヌクレイノパチーとして総称される神経変性疾患の原因となる。
α-シヌクレインの凝集と細胞死を引き起こす機序として、Arf6によるPIP5Kγの活性化の関与が報告されている[110]。初代神経細胞において、家族性パーキンソン病の原因となる変異を持つα-シヌクレイン(A53T)の発現やα-シヌクレインフィブリルを暴露することにより、PIP5KγがArf6依存的に細胞膜にリクルートされPI(4,5)P2の産生が促進され、その結果、α-シヌクレインのさらなる凝集とPI(4,5)P2代謝産物であるイノシトール3リン酸(IP3)の増加によるIP3受容体を介した細胞内Ca2+の増加が引き起こされる。さらに変異α-シヌクレイン(A53T)やα-シヌクレインフィブリルは、小胞体のIP3受容体とミトコンドリアの電位依存性アニオンチャンネルとのカップリングを促し、ミトコンドリア内でのCa2+の上昇と活性酸素種の生成が促進され、細胞死が引き起こされるモデルが提唱されている[110]。
末梢神経障害(軸索型ニューロパチー)
クラスIとクラスII Arfに対するGEFであるGBF1の遺伝子変異が顕性遺伝性ニューロパチーを示す4家系の患者から見出されている[111]。その臨床像は多彩で、20~50歳代で発症し、運動神経のみの障害(遺伝性運動ニューロパチー)あるいは運動神経と感覚神経の両方の障害(Charcot-Marie-Tooth病2型)を呈し、主に遠位筋の筋力低下と萎縮を伴う。4家系から見出された4種類の遺伝子変異の部位は主にGBF1のゴルジ装置への局在に関わるHDB (homology downstream of the Sec7) 1領域とHDB2領域に存在し、患者から樹立した線維芽細胞株はゴルジ装置の細胞質全体への分散などの構造異常を示す[111]。
参考文献
- ↑
Enomoto, K., & Gill, D.M. (1980).
Cholera toxin activation of adenylate cyclase. Roles of nucleoside triphosphates and a macromolecular factor in the ADP ribosylation of the GTP-dependent regulatory component. The Journal of biological chemistry, 255(4), 1252-8. [PubMed:6766444] [WorldCat] - ↑
Kahn, R.A., & Gilman, A.G. (1984).
Purification of a protein cofactor required for ADP-ribosylation of the stimulatory regulatory component of adenylate cyclase by cholera toxin. The Journal of biological chemistry, 259(10), 6228-34. [PubMed:6327671] [WorldCat] - ↑
Schleifer, L.S., Kahn, R.A., Hanski, E., Northup, J.K., Sternweis, P.C., & Gilman, A.G. (1982).
Requirements for cholera toxin-dependent ADP-ribosylation of the purified regulatory component of adenylate cyclase. The Journal of biological chemistry, 257(1), 20-3. [PubMed:6273425] [WorldCat] - ↑ 4.0 4.1 4.2
Kahn, R.A., & Gilman, A.G. (1986).
The protein cofactor necessary for ADP-ribosylation of Gs by cholera toxin is itself a GTP binding protein. The Journal of biological chemistry, 261(17), 7906-11. [PubMed:3086320] [WorldCat] - ↑
Sewell, J.L., & Kahn, R.A. (1988).
Sequences of the bovine and yeast ADP-ribosylation factor and comparison to other GTP-binding proteins. Proceedings of the National Academy of Sciences of the United States of America, 85(13), 4620-4. [PubMed:3133654] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1 6.2
Hosaka, M., Toda, K., Takatsu, H., Torii, S., Murakami, K., & Nakayama, K. (1996).
Structure and intracellular localization of mouse ADP-ribosylation factors type 1 to type 6 (ARF1-ARF6). Journal of biochemistry, 120(4), 813-9. [PubMed:8947846] [WorldCat] [DOI] - ↑
Kahn, R.A., Cherfils, J., Elias, M., Lovering, R.C., Munro, S., & Schurmann, A. (2006).
Nomenclature for the human Arf family of GTP-binding proteins: ARF, ARL, and SAR proteins. The Journal of cell biology, 172(5), 645-50. [PubMed:16505163] [PMC] [WorldCat] [DOI] - ↑ 8.0 8.1 8.2 8.3
Gillingham, A.K., & Munro, S. (2007).
The small G proteins of the Arf family and their regulators. Annual review of cell and developmental biology, 23, 579-611. [PubMed:17506703] [WorldCat] [DOI] - ↑
Pasqualato, S., Ménétrey, J., Franco, M., & Cherfils, J. (2001).
The structural GDP/GTP cycle of human Arf6. EMBO reports, 2(3), 234-8. [PubMed:11266366] [PMC] [WorldCat] [DOI] - ↑ 10.0 10.1
D'Souza-Schorey, C., & Chavrier, P. (2006).
ARF proteins: roles in membrane traffic and beyond. Nature reviews. Molecular cell biology, 7(5), 347-58. [PubMed:16633337] [WorldCat] [DOI] - ↑
Donaldson, J.G., & Jackson, C.L. (2011).
ARF family G proteins and their regulators: roles in membrane transport, development and disease. Nature reviews. Molecular cell biology, 12(6), 362-75. [PubMed:21587297] [PMC] [WorldCat] [DOI] - ↑ 12.0 12.1
Cheung, H.N., Dunbar, C., Mórotz, G.M., Cheng, W.H., Chan, H.Y., Miller, C.C., & Lau, K.F. (2014).
FE65 interacts with ADP-ribosylation factor 6 to promote neurite outgrowth. FASEB journal : official publication of the Federation of American Societies for Experimental Biology, 28(1), 337-49. [PubMed:24056087] [PMC] [WorldCat] [DOI] - ↑
Koo, T.H., Eipper, B.A., & Donaldson, J.G. (2007).
Arf6 recruits the Rac GEF Kalirin to the plasma membrane facilitating Rac activation. BMC cell biology, 8, 29. [PubMed:17640372] [PMC] [WorldCat] [DOI] - ↑
Falace, A., Filipello, F., La Padula, V., Vanni, N., Madia, F., De Pietri Tonelli, D., ..., & Zara, F. (2010).
TBC1D24, an ARF6-interacting protein, is mutated in familial infantile myoclonic epilepsy. American journal of human genetics, 87(3), 365-70. [PubMed:20727515] [PMC] [WorldCat] [DOI] - ↑
Radhakrishna, H., & Donaldson, J.G. (1997).
ADP-ribosylation factor 6 regulates a novel plasma membrane recycling pathway. The Journal of cell biology, 139(1), 49-61. [PubMed:9314528] [PMC] [WorldCat] [DOI] - ↑
Wong-Dilworth, L., Rodilla-Ramirez, C., Fox, E., Restel, S.D., Stockhammer, A., Adarska, P., & Bottanelli, F. (2023).
STED imaging of endogenously tagged ARF GTPases reveals their distinct nanoscale localizations. The Journal of cell biology, 222(7). [PubMed:37102998] [PMC] [WorldCat] [DOI] - ↑
Chardin, P., Paris, S., Antonny, B., Robineau, S., Béraud-Dufour, S., Jackson, C.L., & Chabre, M. (1996).
A human exchange factor for ARF contains Sec7- and pleckstrin-homology domains. Nature, 384(6608), 481-4. [PubMed:8945478] [WorldCat] [DOI] - ↑
Peyroche, A., Paris, S., & Jackson, C.L. (1996).
Nucleotide exchange on ARF mediated by yeast Gea1 protein. Nature, 384(6608), 479-81. [PubMed:8945477] [WorldCat] [DOI] - ↑ 19.0 19.1
Cox, R., Mason-Gamer, R.J., Jackson, C.L., & Segev, N. (2004).
Phylogenetic analysis of Sec7-domain-containing Arf nucleotide exchangers. Molecular biology of the cell, 15(4), 1487-505. [PubMed:14742722] [PMC] [WorldCat] [DOI] - ↑ 20.0 20.1
Sztul, E., Chen, P.W., Casanova, J.E., Cherfils, J., Dacks, J.B., Lambright, D.G., ..., & Kahn, R.A. (2019).
ARF GTPases and their GEFs and GAPs: concepts and challenges. Molecular biology of the cell, 30(11), 1249-1271. [PubMed:31084567] [PMC] [WorldCat] [DOI] - ↑
Yano, H., Kobayashi, I., Onodera, Y., Luton, F., Franco, M., Mazaki, Y., ..., & Sabe, H. (2008).
Fbx8 makes Arf6 refractory to function via ubiquitination. Molecular biology of the cell, 19(3), 822-32. [PubMed:18094045] [PMC] [WorldCat] [DOI] - ↑
Fukaya, M., Sugawara, T., Hara, Y., Itakura, M., Watanabe, M., & Sakagami, H. (2020).
BRAG2a Mediates mGluR-Dependent AMPA Receptor Internalization at Excitatory Postsynapses through the Interaction with PSD-95 and Endophilin 3. The Journal of neuroscience : the official journal of the Society for Neuroscience, 40(22), 4277-4296. [PubMed:32341099] [PMC] [WorldCat] [DOI] - ↑
Sakagami, H., Sanda, M., Fukaya, M., Miyazaki, T., Sukegawa, J., Yanagisawa, T., ..., & Kondo, H. (2008).
IQ-ArfGEF/BRAG1 is a guanine nucleotide exchange factor for Arf6 that interacts with PSD-95 at postsynaptic density of excitatory synapses. Neuroscience research, 60(2), 199-212. [PubMed:18164504] [WorldCat] [DOI] - ↑
Ito, A., Fukaya, M., Sugawara, T., Hara, Y., Okamoto, H., Yamauchi, J., & Sakagami, H. (2021).
Cytohesin-2 mediates group I metabotropic glutamate receptor-dependent mechanical allodynia through the activation of ADP ribosylation factor 6 in the spinal cord. Neurobiology of disease, 159, 105466. [PubMed:34390832] [WorldCat] [DOI] - ↑
Kitano, J., Kimura, K., Yamazaki, Y., Soda, T., Shigemoto, R., Nakajima, Y., & Nakanishi, S. (2002).
Tamalin, a PDZ domain-containing protein, links a protein complex formation of group 1 metabotropic glutamate receptors and the guanine nucleotide exchange factor cytohesins. The Journal of neuroscience : the official journal of the Society for Neuroscience, 22(4), 1280-9. [PubMed:11850456] [PMC] [WorldCat] [DOI] - ↑
Fukaya, M., Kamata, A., Hara, Y., Tamaki, H., Katsumata, O., Ito, N., ..., & Sakagami, H. (2011).
SynArfGEF is a guanine nucleotide exchange factor for Arf6 and localizes preferentially at post-synaptic specializations of inhibitory synapses. Journal of neurochemistry, 116(6), 1122-37. [PubMed:21198641] [WorldCat] [DOI] - ↑
Um, J.W., Choii, G., Park, D., Kim, D., Jeon, S., Kang, H., ..., & Ko, J. (2016).
IQ Motif and SEC7 Domain-containing Protein 3 (IQSEC3) Interacts with Gephyrin to Promote Inhibitory Synapse Formation. The Journal of biological chemistry, 291(19), 10119-30. [PubMed:27002143] [PMC] [WorldCat] [DOI] - ↑
Randazzo, P.A., & Kahn, R.A. (1994).
GTP hydrolysis by ADP-ribosylation factor is dependent on both an ADP-ribosylation factor GTPase-activating protein and acid phospholipids. The Journal of biological chemistry, 269(14), 10758-63. [PubMed:8144664] [WorldCat] - ↑
Makler, V., Cukierman, E., Rotman, M., Admon, A., & Cassel, D. (1995).
ADP-ribosylation factor-directed GTPase-activating protein. Purification and partial characterization. The Journal of biological chemistry, 270(10), 5232-7. [PubMed:7890632] [WorldCat] [DOI] - ↑
Cukierman, E., Huber, I., Rotman, M., & Cassel, D. (1995).
The ARF1 GTPase-activating protein: zinc finger motif and Golgi complex localization. Science (New York, N.Y.), 270(5244), 1999-2002. [PubMed:8533093] [WorldCat] [DOI] - ↑ 31.0 31.1
Kahn, R.A., Bruford, E., Inoue, H., Logsdon, J.M., Nie, Z., Premont, R.T., ..., & Cassel, D. (2008).
Consensus nomenclature for the human ArfGAP domain-containing proteins. The Journal of cell biology, 182(6), 1039-44. [PubMed:18809720] [PMC] [WorldCat] [DOI] - ↑
Miura, K., Jacques, K.M., Stauffer, S., Kubosaki, A., Zhu, K., Hirsch, D.S., ..., & Randazzo, P.A. (2002).
ARAP1: a point of convergence for Arf and Rho signaling. Molecular cell, 9(1), 109-19. [PubMed:11804590] [WorldCat] [DOI] - ↑
Zhao, Z.S., Manser, E., Loo, T.H., & Lim, L. (2000).
Coupling of PAK-interacting exchange factor PIX to GIT1 promotes focal complex disassembly. Molecular and cellular biology, 20(17), 6354-63. [PubMed:10938112] [PMC] [WorldCat] [DOI] - ↑
East, M.P., Bowzard, J.B., Dacks, J.B., & Kahn, R.A. (2012).
ELMO domains, evolutionary and functional characterization of a novel GTPase-activating protein (GAP) domain for Arf protein family GTPases. The Journal of biological chemistry, 287(47), 39538-53. [PubMed:23014990] [PMC] [WorldCat] [DOI] - ↑
Ivanova, A.A., East, M.P., Yi, S.L., & Kahn, R.A. (2014).
Characterization of recombinant ELMOD (cell engulfment and motility domain) proteins as GTPase-activating proteins (GAPs) for ARF family GTPases. The Journal of biological chemistry, 289(16), 11111-11121. [PubMed:24616099] [PMC] [WorldCat] [DOI] - ↑ 36.0 36.1
Su, M.Y., Fromm, S.A., Remis, J., Toso, D.B., & Hurley, J.H. (2021).
Structural basis for the ARF GAP activity and specificity of the C9orf72 complex. Nature communications, 12(1), 3786. [PubMed:34145292] [PMC] [WorldCat] [DOI] - ↑ 37.0 37.1
Su, M.Y., Fromm, S.A., Zoncu, R., & Hurley, J.H. (2020).
Structure of the C9orf72 ARF GAP complex that is haploinsufficient in ALS and FTD. Nature, 585(7824), 251-255. [PubMed:32848248] [PMC] [WorldCat] [DOI] - ↑
Hayakawa, N., Ogoh, H., Sumiyoshi, M., Matsui, Y., Nishikawa, S., Miyamoto, K., ..., & Watanabe, T. (2014).
The ADP-ribosylation factor 1 gene is indispensable for mouse embryonic development after implantation. Biochemical and biophysical research communications, 453(4), 748-53. [PubMed:25305484] [WorldCat] [DOI] - ↑
Follit, J.A., San Agustin, J.T., Jonassen, J.A., Huang, T., Rivera-Perez, J.A., Tremblay, K.D., & Pazour, G.J. (2014).
Arf4 is required for Mammalian development but dispensable for ciliary assembly. PLoS genetics, 10(2), e1004170. [PubMed:24586199] [PMC] [WorldCat] [DOI] - ↑ 40.0 40.1
Hosoi, N., Shibasaki, K., Hosono, M., Konno, A., Shinoda, Y., Kiyonari, H., ..., & Sadakata, T. (2019).
Deletion of Class II ADP-Ribosylation Factors in Mice Causes Tremor by the Nav1.6 Loss in Cerebellar Purkinje Cell Axon Initial Segments. The Journal of neuroscience : the official journal of the Society for Neuroscience, 39(32), 6339-6353. [PubMed:31201232] [PMC] [WorldCat] [DOI] - ↑
Suzuki, T., Kanai, Y., Hara, T., Sasaki, J., Sasaki, T., Kohara, M., ..., & Kanaho, Y. (2006).
Crucial role of the small GTPase ARF6 in hepatic cord formation during liver development. Molecular and cellular biology, 26(16), 6149-56. [PubMed:16880525] [PMC] [WorldCat] [DOI] - ↑ 42.0 42.1 42.2
Kinoshita-Kawada, M., Hasegawa, H., Hongu, T., Yanagi, S., Kanaho, Y., Masai, I., ..., & Wu, J.Y. (2019).
A crucial role for Arf6 in the response of commissural axons to Slit. Development (Cambridge, England), 146(3). [PubMed:30674481] [PMC] [WorldCat] [DOI] - ↑ 43.0 43.1 43.2
Sheen, V.L., Ganesh, V.S., Topcu, M., Sebire, G., Bodell, A., Hill, R.S., ..., & Walsh, C.A. (2004).
Mutations in ARFGEF2 implicate vesicle trafficking in neural progenitor proliferation and migration in the human cerebral cortex. Nature genetics, 36(1), 69-76. [PubMed:14647276] [WorldCat] [DOI] - ↑
Ferland, R.J., Batiz, L.F., Neal, J., Lian, G., Bundock, E., Lu, J., ..., & Sheen, V.L. (2009).
Disruption of neural progenitors along the ventricular and subventricular zones in periventricular heterotopia. Human molecular genetics, 18(3), 497-516. [PubMed:18996916] [PMC] [WorldCat] [DOI] - ↑
Zhang, J., Neal, J., Lian, G., Shi, B., Ferland, R.J., & Sheen, V. (2012).
Brefeldin A-inhibited guanine exchange factor 2 regulates filamin A phosphorylation and neuronal migration. The Journal of neuroscience : the official journal of the Society for Neuroscience, 32(36), 12619-29. [PubMed:22956851] [PMC] [WorldCat] [DOI] - ↑ 46.0 46.1 46.2
Ishida, M., Otero, M.G., Freeman, C., Sánchez-Lara, P.A., Guardia, C.M., Pierson, T.M., & Bonifacino, J.S. (2023).
A neurodevelopmental disorder associated with an activating de novo missense variant in ARF1. Human molecular genetics, 32(7), 1162-1174. [PubMed:36345169] [PMC] [WorldCat] [DOI] - ↑
Arvanitis, D.N., Béhar, A., Tryoen-Tóth, P., Bush, J.O., Jungas, T., Vitale, N., & Davy, A. (2013).
Ephrin B1 maintains apical adhesion of neural progenitors. Development (Cambridge, England), 140(10), 2082-92. [PubMed:23578932] [WorldCat] [DOI] - ↑ 48.0 48.1
Falace, A., Buhler, E., Fadda, M., Watrin, F., Lippiello, P., Pallesi-Pocachard, E., ..., & Cardoso, C. (2014).
TBC1D24 regulates neuronal migration and maturation through modulation of the ARF6-dependent pathway. Proceedings of the National Academy of Sciences of the United States of America, 111(6), 2337-42. [PubMed:24469796] [PMC] [WorldCat] [DOI] - ↑ 49.0 49.1 49.2
Hara, Y., Fukaya, M., Hayashi, K., Kawauchi, T., Nakajima, K., & Sakagami, H. (2016).
ADP Ribosylation Factor 6 Regulates Neuronal Migration in the Developing Cerebral Cortex through FIP3/Arfophilin-1-dependent Endosomal Trafficking of N-cadherin. eNeuro, 3(4). [PubMed:27622210] [PMC] [WorldCat] [DOI] - ↑ 50.0 50.1 50.2
Hara, Y., Katsuyama, T., Fukaya, M., Sugawara, T., Shiroshima, T., Sadakata, T., ..., & Sakagami, H. (2023).
ADP Ribosylation Factor 4 (Arf4) Regulates Radial Migration through N-Cadherin Trafficking during Cerebral Cortical Development. eNeuro, 10(11). [PubMed:37848288] [PMC] [WorldCat] [DOI] - ↑ 51.0 51.1
Ishiguro, T., Noda, M., Nishikawa, M., Nagata, K.I., & Ito, H. (2024).
Variations associated with neurodevelopmental disorders affect ARF1 function and cortical development. Journal of biochemistry, 176(5), 347-357. [PubMed:39052890] [WorldCat] [DOI] - ↑
Miura, Y., & Kanaho, Y. (2017).
ACAP3, the GTPase-activating protein specific to the small GTPase Arf6, regulates neuronal migration in the developing cerebral cortex. Biochemical and biophysical research communications, 493(2), 1089-1094. [PubMed:28919417] [WorldCat] [DOI] - ↑ 53.0 53.1
Hernández-Deviez, D.J., Roth, M.G., Casanova, J.E., & Wilson, J.M. (2004).
ARNO and ARF6 regulate axonal elongation and branching through downstream activation of phosphatidylinositol 4-phosphate 5-kinase alpha. Molecular biology of the cell, 15(1), 111-20. [PubMed:14565977] [PMC] [WorldCat] [DOI] - ↑ 54.0 54.1
Eva, R., Crisp, S., Marland, J.R., Norman, J.C., Kanamarlapudi, V., ffrench-Constant, C., & Fawcett, J.W. (2012).
ARF6 directs axon transport and traffic of integrins and regulates axon growth in adult DRG neurons. The Journal of neuroscience : the official journal of the Society for Neuroscience, 32(30), 10352-64. [PubMed:22836268] [PMC] [WorldCat] [DOI] - ↑
Onishi, K., Shafer, B., Lo, C., Tissir, F., Goffinet, A.M., & Zou, Y. (2013).
Antagonistic functions of Dishevelleds regulate Frizzled3 endocytosis via filopodia tips in Wnt-mediated growth cone guidance. The Journal of neuroscience : the official journal of the Society for Neuroscience, 33(49), 19071-85. [PubMed:24305805] [PMC] [WorldCat] [DOI] - ↑
Onel, S., Bolke, L., & Klämbt, C. (2004).
The Drosophila ARF6-GEF Schizo controls commissure formation by regulating Slit. Development (Cambridge, England), 131(11), 2587-94. [PubMed:15148300] [WorldCat] [DOI] - ↑
Cason, S.E., & Holzbaur, E.L.F. (2023).
Axonal transport of autophagosomes is regulated by dynein activators JIP3/JIP4 and ARF/RAB GTPases. The Journal of cell biology, 222(12). [PubMed:37909920] [PMC] [WorldCat] [DOI] - ↑
Eva, R., Koseki, H., Kanamarlapudi, V., & Fawcett, J.W. (2017).
EFA6 regulates selective polarised transport and axon regeneration from the axon initial segment. Journal of cell science, 130(21), 3663-3675. [PubMed:28935671] [PMC] [WorldCat] [DOI] - ↑
Hong, E.H., Kim, J.Y., Kim, J.H., Lim, D.S., Kim, M., & Kim, J.Y. (2018).
BIG2-ARF1-RhoA-mDia1 Signaling Regulates Dendritic Golgi Polarization in Hippocampal Neurons. Molecular neurobiology, 55(10), 7701-7716. [PubMed:29455446] [WorldCat] [DOI] - ↑ 60.0 60.1
Han, J.S., Hino, K., Li, W., Reyes, R.V., Canales, C.P., Miltner, A.M., ..., & Simó, S. (2020).
CRL5-dependent regulation of the small GTPases ARL4C and ARF6 controls hippocampal morphogenesis. Proceedings of the National Academy of Sciences of the United States of America, 117(37), 23073-23084. [PubMed:32873638] [PMC] [WorldCat] [DOI] - ↑ 61.0 61.1
Hernández-Deviez, D.J., Casanova, J.E., & Wilson, J.M. (2002).
Regulation of dendritic development by the ARF exchange factor ARNO. Nature neuroscience, 5(7), 623-4. [PubMed:12032543] [WorldCat] [DOI] - ↑
Sakagami, H., Matsuya, S., Nishimura, H., Suzuki, R., & Kondo, H. (2004).
Somatodendritic localization of the mRNA for EFA6A, a guanine nucleotide exchange protein for ARF6, in rat hippocampus and its involvement in dendritic formation. The European journal of neuroscience, 19(4), 863-70. [PubMed:15009133] [WorldCat] [DOI] - ↑
Moore, C.D., Thacker, E.E., Larimore, J., Gaston, D., Underwood, A., Kearns, B., ..., & Theibert, A. (2007).
The neuronal Arf GAP centaurin alpha1 modulates dendritic differentiation. Journal of cell science, 120(Pt 15), 2683-93. [PubMed:17635995] [PMC] [WorldCat] [DOI] - ↑
Miura, Y., Hongu, T., Yamauchi, Y., Funakoshi, Y., Katagiri, N., Ohbayashi, N., & Kanaho, Y. (2016).
ACAP3 regulates neurite outgrowth through its GAP activity specific to Arf6 in mouse hippocampal neurons. The Biochemical journal, 473(17), 2591-602. [PubMed:27330119] [WorldCat] [DOI] - ↑
Suzuki, A., Arikawa, C., Kuwahara, Y., Itoh, K., Watanabe, M., Watanabe, H., ..., & Kanaho, Y. (2010).
The scaffold protein JIP3 functions as a downstream effector of the small GTPase ARF6 to regulate neurite morphogenesis of cortical neurons. FEBS letters, 584(13), 2801-6. [PubMed:20493856] [WorldCat] [DOI] - ↑
Yazaki, Y., Hara, Y., Tamaki, H., Fukaya, M., & Sakagami, H. (2014).
Endosomal localization of FIP3/Arfophilin-1 and its involvement in dendritic formation of mouse hippocampal neurons. Brain research, 1557, 55-65. [PubMed:24576489] [WorldCat] [DOI] - ↑ 67.0 67.1 67.2
Rocca, D.L., Amici, M., Antoniou, A., Blanco Suarez, E., Halemani, N., Murk, K., ..., & Hanley, J.G. (2013).
The small GTPase Arf1 modulates Arp2/3-mediated actin polymerization via PICK1 to regulate synaptic plasticity. Neuron, 79(2), 293-307. [PubMed:23889934] [PMC] [WorldCat] [DOI] - ↑ 68.0 68.1 68.2
Choi, S., Ko, J., Lee, J.R., Lee, H.W., Kim, K., Chung, H.S., ..., & Kim, E. (2006).
ARF6 and EFA6A regulate the development and maintenance of dendritic spines. The Journal of neuroscience : the official journal of the Society for Neuroscience, 26(18), 4811-9. [PubMed:16672654] [PMC] [WorldCat] [DOI] - ↑ 69.0 69.1 69.2 69.3
Kim, Y., Lee, S.E., Park, J., Kim, M., Lee, B., Hwang, D., & Chang, S. (2015).
ADP-ribosylation factor 6 (ARF6) bidirectionally regulates dendritic spine formation depending on neuronal maturation and activity. The Journal of biological chemistry, 290(12), 7323-35. [PubMed:25605715] [PMC] [WorldCat] [DOI] - ↑ 70.0 70.1
Miyazaki, H., Yamazaki, M., Watanabe, H., Maehama, T., Yokozeki, T., & Kanaho, Y. (2005).
The small GTPase ADP-ribosylation factor 6 negatively regulates dendritic spine formation. FEBS letters, 579(30), 6834-8. [PubMed:16325184] [WorldCat] [DOI] - ↑ 71.0 71.1
Raemaekers, T., Peric, A., Baatsen, P., Sannerud, R., Declerck, I., Baert, V., ..., & Annaert, W. (2012).
ARF6-mediated endosomal transport of Telencephalin affects dendritic filopodia-to-spine maturation. The EMBO journal, 31(15), 3252-69. [PubMed:22781129] [PMC] [WorldCat] [DOI] - ↑
Lin, L., Lyu, Q., Kwan, P.Y., Zhao, J., Fan, R., Chai, A., ..., & Lai, K.O. (2020).
The epilepsy and intellectual disability-associated protein TBC1D24 regulates the maintenance of excitatory synapses and animal behaviors. PLoS genetics, 16(1), e1008587. [PubMed:32004315] [PMC] [WorldCat] [DOI] - ↑ 73.0 73.1
Ansar, M., Chung, H.L., Al-Otaibi, A., Elagabani, M.N., Ravenscroft, T.A., Paracha, S.A., ..., & Antonarakis, S.E. (2019).
Bi-allelic Variants in IQSEC1 Cause Intellectual Disability, Developmental Delay, and Short Stature. American journal of human genetics, 105(5), 907-920. [PubMed:31607425] [PMC] [WorldCat] [DOI] - ↑
Zhang, H., Webb, D.J., Asmussen, H., Niu, S., & Horwitz, A.F. (2005).
A GIT1/PIX/Rac/PAK signaling module regulates spine morphogenesis and synapse formation through MLC. The Journal of neuroscience : the official journal of the Society for Neuroscience, 25(13), 3379-88. [PubMed:15800193] [PMC] [WorldCat] [DOI] - ↑
Arnold, M., Cross, R., Singleton, K.S., Zlatic, S., Chapleau, C., Mullin, A.P., ..., & Larimore, J. (2016).
The Endosome Localized Arf-GAP AGAP1 Modulates Dendritic Spine Morphology Downstream of the Neurodevelopmental Disorder Factor Dysbindin. Frontiers in cellular neuroscience, 10, 218. [PubMed:27713690] [PMC] [WorldCat] [DOI] - ↑ 76.0 76.1
Szatmari, E.M., Moran, C., Cohen, S., Jacob, A., Parra-Bueno, P., Kamasawa, N., ..., & Yasuda, R. (2021).
ADAP1/Centaurin-α1 Negatively Regulates Dendritic Spine Function and Memory Formation in the Hippocampus. eNeuro, 8(1). [PubMed:33139322] [PMC] [WorldCat] [DOI] - ↑
Ashery, U., Koch, H., Scheuss, V., Brose, N., & Rettig, J. (1999).
A presynaptic role for the ADP ribosylation factor (ARF)-specific GDP/GTP exchange factor msec7-1. Proceedings of the National Academy of Sciences of the United States of America, 96(3), 1094-9. [PubMed:9927699] [PMC] [WorldCat] [DOI] - ↑
Krauss, M., Kinuta, M., Wenk, M.R., De Camilli, P., Takei, K., & Haucke, V. (2003).
ARF6 stimulates clathrin/AP-2 recruitment to synaptic membranes by activating phosphatidylinositol phosphate kinase type Igamma. The Journal of cell biology, 162(1), 113-24. [PubMed:12847086] [PMC] [WorldCat] [DOI] - ↑ 79.0 79.1
Tagliatti, E., Fadda, M., Falace, A., Benfenati, F., & Fassio, A. (2016).
Arf6 regulates the cycling and the readily releasable pool of synaptic vesicles at hippocampal synapse. eLife, 5. [PubMed:26731518] [PMC] [WorldCat] [DOI] - ↑
Kim, S., Ko, J., Shin, H., Lee, J.R., Lim, C., Han, J.H., ..., & Kim, E. (2003).
The GIT family of proteins forms multimers and associates with the presynaptic cytomatrix protein Piccolo. The Journal of biological chemistry, 278(8), 6291-300. [PubMed:12473661] [WorldCat] [DOI] - ↑
Montesinos, M.S., Dong, W., Goff, K., Das, B., Guerrero-Given, D., Schmalzigaug, R., ..., & Young, S.M. (2015).
Presynaptic Deletion of GIT Proteins Results in Increased Synaptic Strength at a Mammalian Central Synapse. Neuron, 88(5), 918-925. [PubMed:26637799] [PMC] [WorldCat] [DOI] - ↑
Faúndez, V., Horng, J.T., & Kelly, R.B. (1997).
ADP ribosylation factor 1 is required for synaptic vesicle budding in PC12 cells. The Journal of cell biology, 138(3), 505-15. [PubMed:9245782] [PMC] [WorldCat] [DOI] - ↑ 83.0 83.1 83.2
Scholz, R., Berberich, S., Rathgeber, L., Kolleker, A., Köhr, G., & Kornau, H.C. (2010).
AMPA receptor signaling through BRAG2 and Arf6 critical for long-term synaptic depression. Neuron, 66(5), 768-80. [PubMed:20547133] [WorldCat] [DOI] - ↑
Broutman, G., & Baudry, M. (2001).
Involvement of the secretory pathway for AMPA receptors in NMDA-induced potentiation in hippocampus. The Journal of neuroscience : the official journal of the Society for Neuroscience, 21(1), 27-34. [PubMed:11150316] [PMC] [WorldCat] [DOI] - ↑
Oku, Y., & Huganir, R.L. (2013).
AGAP3 and Arf6 regulate trafficking of AMPA receptors and synaptic plasticity. The Journal of neuroscience : the official journal of the Society for Neuroscience, 33(31), 12586-98. [PubMed:23904596] [PMC] [WorldCat] [DOI] - ↑
Torii, T., Miyamoto, Y., & Yamauchi, J. (2024).
Myelination by signaling through Arf guanine nucleotide exchange factor. Journal of neurochemistry, 168(9), 2201-2213. [PubMed:38894552] [WorldCat] [DOI] - ↑
Miyamoto, Y., Torii, T., Homma, K., Oizumi, H., Ohbuchi, K., Mizoguchi, K., ..., & Yamauchi, J. (2022).
The adaptor SH2B1 and the phosphatase PTP4A1 regulate the phosphorylation of cytohesin-2 in myelinating Schwann cells in mice. Science signaling, 15(718), eabi5276. [PubMed:35077201] [WorldCat] [DOI] - ↑ 88.0 88.1 88.2
Yamauchi, J., Miyamoto, Y., Torii, T., Takashima, S., Kondo, K., Kawahara, K., ..., & Tanoue, A. (2012).
Phosphorylation of cytohesin-1 by Fyn is required for initiation of myelination and the extent of myelination during development. Science signaling, 5(243), ra69. [PubMed:23012656] [WorldCat] [DOI] - ↑
Torii, T., Ohno, N., Miyamoto, Y., Kawahara, K., Saitoh, Y., Nakamura, K., ..., & Yamauchi, J. (2015).
Arf6 guanine-nucleotide exchange factor cytohesin-2 regulates myelination in nerves. Biochemical and biophysical research communications, 460(3), 819-25. [PubMed:25824033] [WorldCat] [DOI] - ↑
Miyamoto, Y., Torii, T., Tago, K., Tanoue, A., Takashima, S., & Yamauchi, J. (2018).
BIG1/Arfgef1 and Arf1 regulate the initiation of myelination by Schwann cells in mice. Science advances, 4(4), eaar4471. [PubMed:29740613] [PMC] [WorldCat] [DOI] - ↑
Miyamoto, Y., Yamamori, N., Torii, T., Tanoue, A., & Yamauchi, J. (2014).
Rab35, acting through ACAP2 switching off Arf6, negatively regulates oligodendrocyte differentiation and myelination. Molecular biology of the cell, 25(9), 1532-42. [PubMed:24600047] [PMC] [WorldCat] [DOI] - ↑
Akiyama, M., Hasegawa, H., Hongu, T., Frohman, M.A., Harada, A., Sakagami, H., & Kanaho, Y. (2014).
Trans-regulation of oligodendrocyte myelination by neurons through small GTPase Arf6-regulated secretion of fibroblast growth factor-2. Nature communications, 5, 4744. [PubMed:25144208] [WorldCat] [DOI] - ↑
Ge, X., Gong, H., Dumas, K., Litwin, J., Phillips, J.J., Waisfisz, Q., ..., & Shieh, J.T. (2016).
Missense-depleted regions in population exomes implicate ras superfamily nucleotide-binding protein alteration in patients with brain malformation. NPJ genomic medicine, 1, 16036-. [PubMed:28868155] [PMC] [WorldCat] [DOI] - ↑
de Sainte Agathe, J.M., Pode-Shakked, B., Naudion, S., Michaud, V., Arveiler, B., Fergelot, P., ..., & Trimouille, A. (2023).
ARF1-related disorder: phenotypic and molecular spectrum. Journal of medical genetics, 60(10), 999-1005. [PubMed:37185208] [PMC] [WorldCat] [DOI] - ↑
Sakamoto, M., Sasaki, K., Sugie, A., Nitta, Y., Kimura, T., Gürsoy, S., ..., & Matsumoto, N. (2021).
De novo ARF3 variants cause neurodevelopmental disorder with brain abnormality. Human molecular genetics, 31(1), 69-81. [PubMed:34346499] [WorldCat] [DOI] - ↑
Morleo, M., Iaconis, D., Chitayat, D., Peluso, I., Marzella, R., Renieri, A., ..., & Franco, B. (2008).
Disruption of the IQSEC2 transcript in a female with X;autosome translocation t(X;20)(p11.2;q11.2) and a phenotype resembling X-linked infantile spasms (ISSX) syndrome. Molecular medicine reports, 1(1), 33-9. [PubMed:21479374] [WorldCat] - ↑
Shoubridge, C., Tarpey, P.S., Abidi, F., Ramsden, S.L., Rujirabanjerd, S., Murphy, J.A., ..., & Gécz, J. (2010).
Mutations in the guanine nucleotide exchange factor gene IQSEC2 cause nonsyndromic intellectual disability. Nature genetics, 42(6), 486-8. [PubMed:20473311] [PMC] [WorldCat] [DOI] - ↑
Shoubridge, C., Harvey, R.J., & Dudding-Byth, T. (2019).
IQSEC2 mutation update and review of the female-specific phenotype spectrum including intellectual disability and epilepsy. Human mutation, 40(1), 5-24. [PubMed:30328660] [WorldCat] [DOI] - ↑
Monies, D., Abouelhoda, M., Assoum, M., Moghrabi, N., Rafiullah, R., Almontashiri, N., ..., & Alkuraya, F.S. (2019).
Lessons Learned from Large-Scale, First-Tier Clinical Exome Sequencing in a Highly Consanguineous Population. American journal of human genetics, 104(6), 1182-1201. [PubMed:31130284] [PMC] [WorldCat] [DOI] - ↑
Sannerud, R., Declerck, I., Peric, A., Raemaekers, T., Menendez, G., Zhou, L., ..., & Annaert, W. (2011).
ADP ribosylation factor 6 (ARF6) controls amyloid precursor protein (APP) processing by mediating the endosomal sorting of BACE1. Proceedings of the National Academy of Sciences of the United States of America, 108(34), E559-68. [PubMed:21825135] [PMC] [WorldCat] [DOI] - ↑
Chia, P.Z., Toh, W.H., Sharples, R., Gasnereau, I., Hill, A.F., & Gleeson, P.A. (2013).
Intracellular itinerary of internalised β-secretase, BACE1, and its potential impact on β-amyloid peptide biogenesis. Traffic (Copenhagen, Denmark), 14(9), 997-1013. [PubMed:23773724] [WorldCat] [DOI] - ↑
Wahle, T., Prager, K., Raffler, N., Haass, C., Famulok, M., & Walter, J. (2005).
GGA proteins regulate retrograde transport of BACE1 from endosomes to the trans-Golgi network. Molecular and cellular neurosciences, 29(3), 453-61. [PubMed:15886016] [WorldCat] [DOI] - ↑ 103.0 103.1
Tang, W., Tam, J.H., Seah, C., Chiu, J., Tyrer, A., Cregan, S.P., ..., & Pasternak, S.H. (2015).
Arf6 controls beta-amyloid production by regulating macropinocytosis of the Amyloid Precursor Protein to lysosomes. Molecular brain, 8, 41. [PubMed:26170135] [PMC] [WorldCat] [DOI] - ↑
Sabo, S.L., Lanier, L.M., Ikin, A.F., Khorkova, O., Sahasrabudhe, S., Greengard, P., & Buxbaum, J.D. (1999).
Regulation of beta-amyloid secretion by FE65, an amyloid protein precursor-binding protein. The Journal of biological chemistry, 274(12), 7952-7. [PubMed:10075692] [WorldCat] [DOI] - ↑
Shrivastava-Ranjan, P., Faundez, V., Fang, G., Rees, H., Lah, J.J., Levey, A.I., & Kahn, R.A. (2008).
Mint3/X11gamma is an ADP-ribosylation factor-dependent adaptor that regulates the traffic of the Alzheimer's Precursor protein from the trans-Golgi network. Molecular biology of the cell, 19(1), 51-64. [PubMed:17959829] [PMC] [WorldCat] [DOI] - ↑
Hill, K., Li, Y., Bennett, M., McKay, M., Zhu, X., Shern, J., ..., & Kahn, R.A. (2003).
Munc18 interacting proteins: ADP-ribosylation factor-dependent coat proteins that regulate the traffic of beta-Alzheimer's precursor protein. The Journal of biological chemistry, 278(38), 36032-40. [PubMed:12842896] [WorldCat] [DOI] - ↑
Tan, J.Z.A., & Gleeson, P.A. (2019).
The role of membrane trafficking in the processing of amyloid precursor protein and production of amyloid peptides in Alzheimer's disease. Biochimica et biophysica acta. Biomembranes, 1861(4), 697-712. [PubMed:30639513] [WorldCat] [DOI] - ↑
Liu, K., Liu, Y., Xu, Y., Nandakumar, K.S., Shen, X., Lin, J., ..., & Zhou, C. (2019).
Regulatory role of Golgi brefeldin A resistance factor-1 in amyloid precursor protein trafficking, cleavage and Aβ formation. Journal of cellular biochemistry, 120(9), 15604-15615. [PubMed:31111546] [WorldCat] [DOI] - ↑ 109.0 109.1 109.2
Sivadasan, R., Hornburg, D., Drepper, C., Frank, N., Jablonka, S., Hansel, A., ..., & Sendtner, M. (2016).
C9ORF72 interaction with cofilin modulates actin dynamics in motor neurons. Nature neuroscience, 19(12), 1610-1618. [PubMed:27723745] [WorldCat] [DOI] - ↑ 110.0 110.1
Horvath, J.D., Casas, M., Kutchukian, C., Sánchez, S.C., Pergande, M.R., Cologna, S.M., ..., & Dickson, E.J. (2023).
α-Synuclein-dependent increases in PIP5K1γ drive inositol signaling to promote neurotoxicity. Cell reports, 42(10), 113244. [PubMed:37838947] [PMC] [WorldCat] [DOI] - ↑ 111.0 111.1
Mendoza-Ferreira, N., Karakaya, M., Cengiz, N., Beijer, D., Brigatti, K.W., Gonzaga-Jauregui, C., ..., & Wirth, B. (2020).
De Novo and Inherited Variants in GBF1 are Associated with Axonal Neuropathy Caused by Golgi Fragmentation. American journal of human genetics, 107(4), 763-777. [PubMed:32937143] [PMC] [WorldCat] [DOI]