「神経型PASドメインタンパク質」の版間の差分
細編集の要約なし |
|||
| (同じ利用者による、間の8版が非表示) | |||
| 31行目: | 31行目: | ||
=== basic Helix-Loop-Helixドメイン === | === basic Helix-Loop-Helixドメイン === | ||
約50アミノ酸からなり、塩基性領域、Helix-Loop-Helix領域2つの機能的部分に分けられる('''図1''')。 | 約50アミノ酸からなり、塩基性領域、Helix-Loop-Helix領域2つの機能的部分に分けられる('''図1''')。 | ||
塩基性領域はドメインのN末端側に位置し、正電荷を持つ[[アミノ酸]]([[リジン]]、[[アルギニン]]など)に富む。[[DNA]]への直接的な結合を担い、標的遺伝子の[[プロモーター]]や[[エンハンサー]]領域に存在する特定のコンセンサス配列、主にE-boxと呼ばれる「CANNTG」(NPASファミリーの場合は特にCACGTGが多い)を認識する<ref name=Murre1989><pubmed>2503252</pubmed></ref><ref name=Yutzey1992><pubmed>1329039</pubmed></ref>('''図2''')。 | 塩基性領域はドメインのN末端側に位置し、正電荷を持つ[[アミノ酸]]([[リジン]]、[[アルギニン]]など)に富む。[[DNA]]への直接的な結合を担い、標的遺伝子の[[プロモーター]]や[[エンハンサー]]領域に存在する特定のコンセンサス配列、主にE-boxと呼ばれる「CANNTG」(NPASファミリーの場合は特にCACGTGが多い)を認識する<ref name=Murre1989><pubmed>2503252</pubmed></ref><ref name=Yutzey1992><pubmed>1329039</pubmed></ref>('''図2''')。 | ||
Helix-Loop-Helix領域は2つの[[αヘリックス]]が柔軟なループ構造で連結された構造。この領域はタンパク質の二量体形成に不可欠である。[[class I bHLH-PASスーパーファミリー]]に属するNPASファミリーはそれぞれ、機能的なDNA結合のために[[class II bHLH-PASスーパーファミリー]]に属する[[ARNT]](別名[[HIF1β]])/[[ARNT2]]、または、その神経系特異的ホモログである[[ARNT-like 1 | Helix-Loop-Helix領域は2つの[[αヘリックス]]が柔軟なループ構造で連結された構造。この領域はタンパク質の二量体形成に不可欠である。[[class I bHLH-PASスーパーファミリー]]に属するNPASファミリーはそれぞれ、機能的なDNA結合のために[[class II bHLH-PASスーパーファミリー]]に属する[[ARNT]](別名[[HIF1β]])/[[ARNT2]]、または、その神経系特異的ホモログである[[ARNTL]]([[ARNT-like 1]]、別名BMAL1/BMAL2と、特異的にヘテロ二量体を形成する<ref name=Swanson1993><pubmed>8287061</pubmed></ref><ref name=Hirose1996><pubmed>8657146</pubmed></ref><ref name=Wu2016 />。 | ||
=== PASドメイン === | === PASドメイン === | ||
| 52行目: | 51行目: | ||
[[転写調節因子]]としての機能を通じて、個体レベルでの多様な生理現象に関与する。 | [[転写調節因子]]としての機能を通じて、個体レベルでの多様な生理現象に関与する。 | ||
=== 転写調節 === | === 転写調節 === | ||
最も基本的な分子機能は、転写因子としての役割である。適切なパートナーと安定なヘテロ二量体を形成した後、標的遺伝子の[[プロモーター]]や[[エンハンサー]]領域に存在する[[E-boxコンセンサス配列]](主にCACGTGまたはその周辺配列)に特異的に結合する<ref name=Wu2016 /><ref name=Sun2022><pubmed>36343253</pubmed></ref>('''図2''')。結合後、C末端領域などを介して[[コアクチベーター]](例:[[サイクリックAMP応答因子結合タンパク質 | 最も基本的な分子機能は、転写因子としての役割である。適切なパートナーと安定なヘテロ二量体を形成した後、標的遺伝子の[[プロモーター]]や[[エンハンサー]]領域に存在する[[E-boxコンセンサス配列]](主にCACGTGまたはその周辺配列)に特異的に結合する<ref name=Wu2016 /><ref name=Sun2022><pubmed>36343253</pubmed></ref>('''図2''')。結合後、C末端領域などを介して[[コアクチベーター]](例:[[サイクリックAMP応答因子結合タンパク質]] ([[cAMP responsive element binding protein-binding protein]], [[CBP]]/[[p300]]); [[nuclear receptor coactivator 1]] ([[NCOA1]])/[[steroid receptor coactivator 1]] ([[SRC-1]]))やコリプレッサー(例: [[nuclear receptor corepressor 2]] ([[NCOR2]])/[[silencing mediator of retinoic acid and thyroid hormone receptor]] ([[SMRT]]))をリクルートすることにより、ヒストンの[[アセチル化]]・[[脱アセチル化]]などの[[クロマチン]]修飾を誘導し、標的遺伝子の転写効率を精密に正または負に制御する<ref name=Bersten2013><pubmed>24263188</pubmed></ref><ref name=Luoma2018><pubmed>30509165</pubmed></ref>。 | ||
=== パートナー選択性と標的遺伝子特異性 === | === パートナー選択性と標的遺伝子特異性 === | ||
| 58行目: | 57行目: | ||
===シグナル伝達経路とのクロストーク === | ===シグナル伝達経路とのクロストーク === | ||
NPASファミリーの活動は、他の細胞内シグナル伝達経路と密接に連携している。例えば、NPAS4の発現は神経活動に伴う[[カルシウム]]流入によって厳密に制御されており <ref name=Lin2008><pubmed>18815592</pubmed></ref>、[[カルシウム/カルモジュリン依存的タンパク質リン酸化酵素]] | NPASファミリーの活動は、他の細胞内シグナル伝達経路と密接に連携している。例えば、NPAS4の発現は神経活動に伴う[[カルシウム]]流入によって厳密に制御されており <ref name=Lin2008><pubmed>18815592</pubmed></ref>、[[カルシウム/カルモジュリン依存的タンパク質リン酸化酵素]]や[[転写因子]] ([[cAMP response element-binding protein]] ([[CREB]]), [[myocyte enhancer factor 2]] ([[MEF2]])がNPAS4遺伝子の発現制御に関与している<ref name=Sun2016><pubmed>26987258</pubmed></ref>。また、NPAS2の活性は概日時計のフィードバックループや代謝産物によって調節される<ref name=Reick2001><pubmed>11441147</pubmed></ref><ref name=Eckel-Mahan2013><pubmed>23303907</pubmed></ref>。このように、NPASファミリーは様々な細胞内外の刺激に応答し、それを転写レベルの変化へと変換する重要な結節点として機能している。 | ||
==NPAS1== | ==NPAS1== | ||
| 73行目: | 72行目: | ||
== NPAS2 == | == NPAS2 == | ||
=== 組織分布 === | === 組織分布 === | ||
中枢神経系では、[[概日リズム]]の中枢である[[視交叉上核]]([[suprachiasmatic nucleus]], [[SCN]])に加えて、大脳皮質、[[海馬]]、[[線条体]]などの[[前脳]]領域で広く発現している<ref name=Reick2001 /> | 中枢神経系では、[[概日リズム]]の中枢である[[視交叉上核]]([[suprachiasmatic nucleus]], [[SCN]])に加えて、大脳皮質、[[海馬]]、[[線条体]]などの[[前脳]]領域で広く発現している<ref name=Reick2001 />(。末梢組織では、[[肝臓]]、[[腎臓]]、肺、[[心臓]]などでも発現が確認されている<ref name=Hogenesch1997><pubmed>9079689</pubmed></ref><ref name=Storch2002><pubmed>11967526</pubmed></ref>。SCNや肝臓においては、その[[mRNA]]およびタンパク質レベルが[[概日周期]]に従ってリズミカルに変動することが知られている<ref name=Reick2001><pubmed>11441147</pubmed></ref><ref name=Storch2002 />。 | ||
=== 機能 === | === 機能 === | ||
==== リガンド応答性 ==== | ==== リガンド応答性 ==== | ||
NPAS2は[[ヘム]]をリガンドとして結合し、細胞内のガス状分子([[CO]], [[酸素|O<sub>2</sub>]], [[NO]])の濃度変化に応じてその立体構造や転写活性が変化する可能性が示唆されている<ref name=Dioum2002><pubmed>12446832</pubmed></ref><ref name=Gilles-Gonzalez2005><pubmed>15598487</pubmed></ref>。これにより、NPAS2は細胞の代謝状態(例:ヘム生合成レベル)やガス環境を感知し、概日リズムや代謝関連遺伝子の発現を調節する役割を担っていると考えられる<ref name=Kitanishi2008><pubmed>18479150</pubmed></ref>。NPAS1, 3, 4も、PAS-Bドメイン内にリガンド結合ポケットを有することが構造的に示されており<ref name=Wu2016 /><ref name=Sun2022><pubmed>36343253</pubmed></ref>('''図2''')、これらのタンパク質は未知の内因性リガンドによって活性が制御されている可能性が考えられる。 | NPAS2は[[ヘム]]をリガンドとして結合し、細胞内のガス状分子([[CO]], [[酸素|O<sub>2</sub>]], [[NO]])の濃度変化に応じてその立体構造や転写活性が変化する可能性が示唆されている<ref name=Dioum2002><pubmed>12446832</pubmed></ref><ref name=Gilles-Gonzalez2005><pubmed>15598487</pubmed></ref>。これにより、NPAS2は細胞の代謝状態(例:ヘム生合成レベル)やガス環境を感知し、概日リズムや代謝関連遺伝子の発現を調節する役割を担っていると考えられる<ref name=Kitanishi2008><pubmed>18479150</pubmed></ref>。NPAS1, 3, 4も、PAS-Bドメイン内にリガンド結合ポケットを有することが構造的に示されており<ref name=Wu2016 /><ref name=Sun2022><pubmed>36343253</pubmed></ref>('''図2''')、これらのタンパク質は未知の内因性リガンドによって活性が制御されている可能性が考えられる。 | ||
==== 概日リズム制御 ==== | ==== 概日リズム制御 ==== | ||
視交叉上核(SCN)において、CLOCKと共にコア[[時計遺伝子]](period ( | 視交叉上核(SCN)において、CLOCKと共にコア[[時計遺伝子]](period (Per)、cryptochrome (CRY)など)の転写を制御する転写活性化因子として機能し、約24時間周期の概日リズム発振に寄与する<ref name=Reick2001><pubmed>11441147</pubmed></ref><ref name=Parekh2019><pubmed>30962277</pubmed></ref>。末梢組織(肝臓、肺など)においても、それぞれの組織における概日時計の維持に関与する<ref name=Storch2002><pubmed>11967526</pubmed></ref>。NPAS2変異マウスは、正常な光周期下では比較的正常な活動リズムを示すが、恒常暗黒下での活動周期の不安定性や、特定の光パルスに対する位相シフト反応の変化、睡眠ホメオスタシスの異常などを示す<ref name=Dudley2003><pubmed>12843397</pubmed></ref><ref name=Mongrain2011><pubmed>22039518</pubmed></ref>。 | ||
==== 代謝調節 ==== | ==== 代謝調節 ==== | ||
特に肝臓において、[[糖新生]]や[[脂質]]代謝に関わる遺伝子の発現を概日的に制御し、[[エネルギー恒常性]]の維持に関与する<ref name=Lee2015><pubmed>25212631</pubmed></ref><ref name=Ma2023><pubmed>36978001</pubmed></ref>。 | 特に肝臓において、[[糖新生]]や[[脂質]]代謝に関わる遺伝子の発現を概日的に制御し、[[エネルギー恒常性]]の維持に関与する<ref name=Lee2015><pubmed>25212631</pubmed></ref><ref name=Ma2023><pubmed>36978001</pubmed></ref>。 | ||
| 113行目: | 112行目: | ||
[[ファイル:Tsuboi NPAS Fig3.png|サムネイル|'''図3. 健常時と損傷時の脳におけるNPAS4の役割'''<br><br> | [[ファイル:Tsuboi NPAS Fig3.png|サムネイル|'''図3. 健常時と損傷時の脳におけるNPAS4の役割'''<br><br> | ||
]] | ]] | ||
[[ファイル:Tsuboi NPAS Fig4.png|サムネイル|'''図4. 神経活動依存的なDNA切断とその修復におけるNPAS4の役割'''<br>文献<ref name=Delint-Ramirez2023><pubmed>37084713</pubmed></ref> | [[ファイル:Tsuboi NPAS Fig4.png|サムネイル|'''図4. 神経活動依存的なDNA切断とその修復におけるNPAS4の役割'''<br>文献<ref name=Delint-Ramirez2023><pubmed>37084713</pubmed></ref>の図を改変)。]] | ||
== NPAS4 == | == NPAS4 == | ||
=== 組織分布 === | === 組織分布 === | ||
| 120行目: | 118行目: | ||
=== 機能 === | === 機能 === | ||
==== 神経活動依存的な遺伝子発現のマスターレギュレーター ==== | ==== 神経活動依存的な遺伝子発現のマスターレギュレーター ==== | ||
NPAS4は、[[興奮性ニューロン]]での標的遺伝子( | NPAS4は、[[興奮性ニューロン]]での標的遺伝子([[BDNF]]など)を活性化させて、[[抑制性シナプス]]の数を増加させることで、回路全体の活動を低下させる一方、抑制性ニューロンでの標的遺伝子([[MDM2]]など)を不活性化させて、[[シナプス形成]]を促進し[[GABA]]の放出を増加させることで、回路全体の活動を低下させるという、[[恒常的な可塑性]]を維持する役割を果たす('''図3''')。 | ||
ニューロンが活動すると迅速に発現が誘導され、その後、抑制性シナプスの形成や機能に関わる遺伝子群([[glutamate decarboxylase 1]]/[[glutamate decarboxylase 2|2]] ([[GAD1]]/[[GAD2|2]]) | ニューロンが活動すると迅速に発現が誘導され、その後、抑制性シナプスの形成や機能に関わる遺伝子群([[glutamate decarboxylase 1]]/[[glutamate decarboxylase 2|2]] ([[GAD1]])/[[GAD2|2]]), [[ソマトスタチン]] ([[SST]])、[[神経栄養因子]]([[brain-derived neurotrophic factor]], [[BDNF]])、[[イオンチャネル]]、その他の転写因子など、多岐にわたる標的遺伝子の発現を協調的に制御する<ref name=Lin2008><pubmed>18815592</pubmed></ref><ref name=Bloodgood2013><pubmed>24201284</pubmed></ref><ref name=Pollina2023><pubmed>36792830</pubmed></ref><ref name=Spiegel2014 />。これにより、神経回路の活動レベルに応じた適応的な変化を引き起こす。 | ||
[[Fos]]、Npas4、[[Egr1]]などの最初期遺伝子(immediately early gene: IEG)のプロモーターでは、感覚刺激により[[topoisomerase IIβ]]([[TOP2B]])を介してDNA二本鎖切断(double-strand break: DSB)が形成される。マウス海馬ニューロンを[[カイニン酸]]で刺激した2時間後に観察されるDSB部位の大部分(69%)は、NPAS4/ARNTヘテロダイマーが最も多く結合しているNpas4遺伝子座と重なっていた<ref name=Pollina2023><pubmed>36792830</pubmed></ref>('''図4''')。 | [[Fos]]、Npas4、[[Egr1]]などの最初期遺伝子(immediately early gene: IEG)のプロモーターでは、感覚刺激により[[topoisomerase IIβ]]([[TOP2B]])を介してDNA二本鎖切断(double-strand break: DSB)が形成される。マウス海馬ニューロンを[[カイニン酸]]で刺激した2時間後に観察されるDSB部位の大部分(69%)は、NPAS4/ARNTヘテロダイマーが最も多く結合しているNpas4遺伝子座と重なっていた<ref name=Pollina2023><pubmed>36792830</pubmed></ref>('''図4''')。 | ||
| 129行目: | 127行目: | ||
==== シナプス可塑性・学習記憶 ==== | ==== シナプス可塑性・学習記憶 ==== | ||
海馬依存的な[[空間記憶]]や[[文脈的恐怖記憶]]、扁桃体依存的な[[手がかり恐怖記憶]]など、様々な種類の学習・記憶課題の遂行に必要である<ref name=Ramamoorthi2011><pubmed>22194569</pubmed></ref><ref name=Ploski2011><pubmed>21887312</pubmed></ref> | 海馬依存的な[[空間記憶]]や[[文脈的恐怖記憶]]、扁桃体依存的な[[手がかり恐怖記憶]]など、様々な種類の学習・記憶課題の遂行に必要である<ref name=Ramamoorthi2011><pubmed>22194569</pubmed></ref><ref name=Ploski2011><pubmed>21887312</pubmed></ref>(。また、[[長期増強]]([[long-term potentiation]], [[LTP]])や[[長期抑圧]]([[long-term depression]], ([[LTD]]))といったシナプス可塑性の誘導や維持にも関与することが示されている<ref name=Bloodgood2013><pubmed>24201284</pubmed></ref>。 | ||
==== 恒常性可塑性と神経保護 ==== | ==== 恒常性可塑性と神経保護 ==== | ||
| 141行目: | 139行目: | ||
=== 疾患との関わり=== | === 疾患との関わり=== | ||
==== てんかん ==== | ==== てんかん ==== | ||
[[てんかん]] | [[てんかん]]発作は強力な神経活動刺激であり、発作時にNPAS4の発現が海馬などで著しく誘導される(Lin et al., 2008)。NPAS4は抑制性シナプスの形成を促進することから、発作後の神経回路の過剰興奮を抑制する保護的な役割(抗てんかん作用)を持つと考えられている。実際に、NPAS4欠損マウスは、薬物誘発性てんかん発作に対する感受性が亢進し、発作重症度が増加することが示されている<ref name=Shan2018><pubmed>29222951</pubmed></ref>。 | ||
==== 脳梗塞・神経変性疾患 ==== | ==== 脳梗塞・神経変性疾患 ==== | ||
脳虚血などの神経傷害後にもNPAS4の発現が誘導され、神経保護に関与する可能性が示唆されている<ref name=Shamloo2006><pubmed>17156197</pubmed></ref><ref name=Takahashi2021><pubmed>34349016</pubmed></ref>('''図3''')。その機能不全が神経変性疾患の病態に関わる可能性も研究されている。 | |||
==== 不安障害・心的外傷後ストレス障害 ==== | ==== 不安障害・心的外傷後ストレス障害 ==== | ||
扁桃体におけるNPAS4の機能は恐怖記憶の形成と消去に関与するため、その調節異常が不安障害や[[心的外傷後ストレス障害]]([[posttraumatic stress disorder]], [[PTSD]])の病態に関与する可能性が考えられている<ref name=Ploski2011><pubmed>21887312</pubmed></ref>。 | |||
==== 依存症 ==== | ==== 依存症 ==== | ||
薬物([[コカイン]]など)投与によって[[線条体]] | 薬物([[コカイン]]など)投与によって[[線条体]]などでNPAS4の発現が誘導され、薬物に対する報酬学習や精神刺激薬感受性、依存からの再燃に関与することが示唆されている<ref name=Taniguchi2017><pubmed>28957664</pubmed></ref>。 | ||
==== 自閉スペクトラム症==== | ==== 自閉スペクトラム症==== | ||
病態仮説の一つである神経回路の興奮/抑制バランスの破綻において、NPAS4が重要な役割を果たしている可能性が注目されている。NPAS4の機能異常とASD病態との関連、および治療標的としての可能性が探求されている<ref name=Rein2021><pubmed>32099100</pubmed></ref>。 | |||
== 関連語 == | == 関連語 == | ||
* [[転写因子]] | * [[転写因子]] | ||
2025年8月13日 (水) 08:11時点における版
坪井 昭夫
大阪大学 大学院薬学研究科神経薬理学
DOI:10.14931/bsd.11087 原稿受付日:2025年5月19日 原稿完成日:2025年5月27日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
英:neuronal PAS domain protein
英略称:NPAS
神経型PASドメインタンパク質 (neuronal PAS domain protein; NPAS)ファミリーはclass I bHLH-PASスーパーファミリーに属する転写因子群であり、4つのメンバーがclass II bHLH-PASスーパーファミリーに属するARNTまたはBMAL1とヘテロ二量体を形成して、E-boxと呼ばれるDNAシスエレメントに結合し標的遺伝子の転写を制御する。これらのタンパク質は主に神経系で発現するが、軟骨形成、概日リズム、神経発生、神経活動に応じた遺伝子発現や記憶といった多様な生理機能にそれぞれ関与しており、その機能不全はがん、睡眠障害、精神疾患、てんかんなど様々な疾患と関連する、神経系の発生・機能・可塑性に重要な分子群である。
神経型PASドメインタンパク質とは
神経型PASドメインタンパク質 (neuronal PAS domain protein; NPAS)ファミリーは、進化的に保存された転写因子の一群であり、その構造的特徴としてN末端側に塩基性ヘリックス・ループ・ヘリックス(basic Helix-Loop-Helix, bHLH)ドメイン、それに続いて2つのPer-ARNT-Sim (PAS)ドメインを持つ[1](図1)。これらのタンパク質は、環境中の化学物質応答(ダイオキシン応答におけるaryl hydrocarbon receptor, AhR)、細胞の低酸素応答(低酸素応答因子, hypoxia-inducible factor, HIF)、概日リズムの制御(circadian locomotor output cycles kaput, CLOCKやbrain and muscle ARNT-like 1, BMAL1)、神経発生など、多様な生物学的プロセスを制御する広範なbHLH-PASスーパーファミリーに属している[2]。PASドメイン自身は約70アミノ酸からなるモジュールであり、タンパク質間相互作用(特に二量体形成)のプラットフォームとして機能するだけでなく、ヘム、フラビン、低分子化合物などの様々なリガンドや補因子を結合することで、外部シグナルや細胞内環境の変化を感知するセンサーとしての役割も担う[3][4]。
NPASファミリーに属する最初のメンバーは、1990年代後半から2000年代初頭にかけて同定された。NPAS1は、低酸素応答転写因子HIFファミリー、特にHIF1αやHIF2αと相同性を持ち、当初はHIF3αのアイソフォームであるinhibitory PAS domain protein (IPAS)として報告された[5][6]。NPAS2(別名members of PAS superfamily 4, MOP4)は、概日リズムの中核因子であるCLOCKとの高い相同性から発見され、哺乳類の概日時計におけるCLOCKの機能的パラログとして同定された[5][7]。その後、NPAS3(別名MOP6)が脳での発現パターンと神経発生における潜在的な役割に基づいてクローニングされ[8][9]、NPAS4(別名MOP8, NXF, Le-PAS)は、神経細胞の活動、特に脱分極やカルシウム流入に応答して迅速かつ一過的に発現が誘導される最初期遺伝子 (immediate early gene, IEG)として、複数の研究グループによって独立に同定された[10][11][12]。
これらの発見とそれに続く研究により、NPASファミリーが神経系の発生、シナプス機能、可塑性、学習・記憶、概日リズム、代謝調節など、極めて多様な生命現象において重要な役割を担っていることが明らかになった。特にNPAS3とNPAS4は神経系での発現が顕著であることから"neuronal" PAS domain proteinと命名された経緯があるが、NPAS1やNPAS2のように神経系以外の組織(肝臓、肺など)での機能も報告されている[13]。
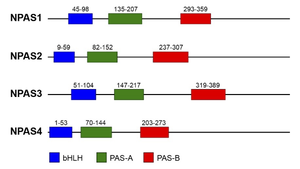
ヒトNPAS1-4タンパク質における3つのドメインの配置図。
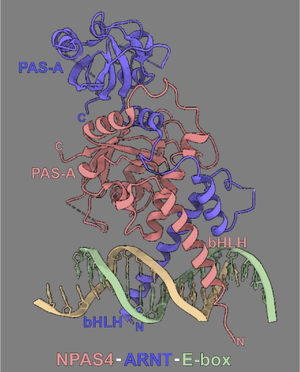
マウスNPAS4-ARNT2-DNA(E-box)複合体の3次元構造の模式図。DNA結合に必要なbHLHドメイン、および、二量体形成に必要なPAS-Aドメインの位置を示す。文献[14]の図を改変。
サブファミリーと構造
哺乳類ではNPASは共通のドメイン構造を持つNPAS1からNPAS4の4つのメンバーによって構成される[1][2](図1)。これらはアミノ酸配列、特にbHLHドメインとPASドメインにおいて高い相同性を示すが、それぞれ異なる遺伝子にコードされ、発現パターンや生理機能、制御機構において独自の特徴を持つ。
NPASは細胞質または核内で、他の転写因子とヘテロ二量体を形成する[15]。複合体として核内に移行、あるいは核内で複合体が形成されると、標的遺伝子の調節領域に存在するE-box配列に結合することで、リクルートした転写共役因子群とともにクロマチン構造の変化やRNAポリメラーゼIIを動員する。その結果、転写を活性化または抑制する。Npas1, 3, 4はARNT/ARNT2と、Npas2はBMAL1/BMAL2と結合し、PASドメイン間の相互作用を介してbHLHドメインによる二量体形成を安定化させる[16](図2)。
4つのNPASメンバーは、これらの基本的な構造と作用機序を共有しつつも、それぞれが異なる時空間的発現パターン、異なる標的遺伝子群、そして異なる生理機能を持つことで、生命現象の多様な側面を分担して制御していると考えられる。
N末端領域
メンバー間で多様性が高く、特定の機能は一概には言えないが、転写活性化ドメインの一部や他のタンパク質との相互作用部位を含むことがある。
basic Helix-Loop-Helixドメイン
約50アミノ酸からなり、塩基性領域、Helix-Loop-Helix領域2つの機能的部分に分けられる(図1)。 塩基性領域はドメインのN末端側に位置し、正電荷を持つアミノ酸(リジン、アルギニンなど)に富む。DNAへの直接的な結合を担い、標的遺伝子のプロモーターやエンハンサー領域に存在する特定のコンセンサス配列、主にE-boxと呼ばれる「CANNTG」(NPASファミリーの場合は特にCACGTGが多い)を認識する[17][18](図2)。
Helix-Loop-Helix領域は2つのαヘリックスが柔軟なループ構造で連結された構造。この領域はタンパク質の二量体形成に不可欠である。class I bHLH-PASスーパーファミリーに属するNPASファミリーはそれぞれ、機能的なDNA結合のためにclass II bHLH-PASスーパーファミリーに属するARNT(別名HIF1β)/ARNT2、または、その神経系特異的ホモログであるARNTL(ARNT-like 1、別名BMAL1/BMAL2と、特異的にヘテロ二量体を形成する[19][20][16]。
PASドメイン
bHLHドメインのC末端側に隣接して、約70アミノ酸からなるPASリピートが2つ(PAS-AとPAS-B)存在する。これらはβシートとαヘリックスからなる特徴的なフォールド構造を形成し、以下の多様な機能を持つ[3][2](図2)。
PASドメイン内部には疎水性のポケット構造が存在し、低分子リガンドや補因子を結合することができる。特にNPAS2は、そのPAS-Aドメインにヘムを共有結合しており、細胞内のガス状分子(一酸化炭素〔CO〕や酸素〔O2〕)の濃度変化を感知するセンサーとして機能し、リガンド結合状態に応じて転写活性が変化する可能性が強く示唆されている[21][22]。NPAS1, 3, 4に関しても、PAS-Bドメイン内にリガンド結合ポケットが存在することが結晶構造解析により明らかにされ、内因性リガンドの存在や、これらのポケットを標的とした低分子化合物による機能制御(創薬標的としての可能性)が期待されている[16][14]。
リガンド結合部位以外にも、他のシグナル伝達分子やシャペロン(例:HSP90)との相互作用部位として機能することが知られている。
C末端領域
PAS-BドメインよりもさらにC末端側の領域は、メンバー間で長さや配列の相同性が低いが、多くの場合、転写活性化ドメイン(transactivation domain)または転写抑制ドメインを含んでいる。この領域を介して、ヒストンアセチル化酵素(histone acetyltransferase, HAT)を含むコアクチベーター複合体や、ヒストン脱アセチル化酵素(histone deacetylase, HDAC)を含むコリプレッサー複合体と相互作用し、標的遺伝子の転写を正または負に調節する[13][23]。
細胞内分布
いずれも転写因子であるため、その主要な機能部位は細胞核内である。細胞質で合成された後、核移行シグナル(nuclear localization signal, NLS)やパートナー分子との結合などによって核内に輸送されると考えられる。NPAS4は、神経活動に応じて発現量がダイナミックに変化し、核内存在量や活性が時間経過と共に厳密に制御されていると考えられる。一部のbHLH-PASタンパク質では、リン酸化などの翻訳後修飾によって核-細胞質間シャトリングが制御される例も知られており[24]、NPASファミリーにおいても同様の制御機構が存在する可能性が考えられるが、詳細なメカニズムはまだ十分に解明されていない。
機能
転写調節因子としての機能を通じて、個体レベルでの多様な生理現象に関与する。
転写調節
最も基本的な分子機能は、転写因子としての役割である。適切なパートナーと安定なヘテロ二量体を形成した後、標的遺伝子のプロモーターやエンハンサー領域に存在するE-boxコンセンサス配列(主にCACGTGまたはその周辺配列)に特異的に結合する[16][14](図2)。結合後、C末端領域などを介してコアクチベーター(例:サイクリックAMP応答因子結合タンパク質 (cAMP responsive element binding protein-binding protein, CBP/p300); nuclear receptor coactivator 1 (NCOA1)/steroid receptor coactivator 1 (SRC-1))やコリプレッサー(例: nuclear receptor corepressor 2 (NCOR2)/silencing mediator of retinoic acid and thyroid hormone receptor (SMRT))をリクルートすることにより、ヒストンのアセチル化・脱アセチル化などのクロマチン修飾を誘導し、標的遺伝子の転写効率を精密に正または負に制御する[13][23]。
パートナー選択性と標的遺伝子特異性
NPAS1, 3, 4はARNT/ARNT2と、NPAS2はBMAL1/BMAL2とヘテロ二量体を形成するが、それぞれのメンバー間における結合親和性や、認識・結合するE-box配列の微妙な違い、あるいはゲノム上の結合部位(プロモーター vs エンハンサー)の選択性が異なる可能性がある。これが、各NPASメンバーが制御する標的遺伝子群の特異性を生み出す一因となっていると考えられる[13][16]。
シグナル伝達経路とのクロストーク
NPASファミリーの活動は、他の細胞内シグナル伝達経路と密接に連携している。例えば、NPAS4の発現は神経活動に伴うカルシウム流入によって厳密に制御されており [25]、カルシウム/カルモジュリン依存的タンパク質リン酸化酵素や転写因子 (cAMP response element-binding protein (CREB), myocyte enhancer factor 2 (MEF2)がNPAS4遺伝子の発現制御に関与している[26]。また、NPAS2の活性は概日時計のフィードバックループや代謝産物によって調節される[7][27]。このように、NPASファミリーは様々な細胞内外の刺激に応答し、それを転写レベルの変化へと変換する重要な結節点として機能している。
NPAS1
組織分布
主に脳と脊髄で発現しており[5][28]、肺などでも検出される[29]。低酸素状態に応答して一部の細胞で発現が誘導されることがある[6]。
機能
神経発生
脳の発生過程において、神経幹細胞の増殖、神経細胞への分化、細胞移動、軸索伸長、および細胞生存を制御する上で極めて重要である[30][28]。
気管形成
気管上皮細胞と間葉系細胞の相互作用に関与し、正常な気管軟骨輪のパターン形成に必須である[31]。
低酸素応答の調節
HIF経路の構成因子として、特にHIF1αやHIF2αの活性を調節(主に抑制)する役割を持つ可能性が、特定の状況下で示唆されている[6]。
NPAS2
組織分布
中枢神経系では、概日リズムの中枢である視交叉上核(suprachiasmatic nucleus, SCN)に加えて、大脳皮質、海馬、線条体などの前脳領域で広く発現している[7](。末梢組織では、肝臓、腎臓、肺、心臓などでも発現が確認されている[5][32]。SCNや肝臓においては、そのmRNAおよびタンパク質レベルが概日周期に従ってリズミカルに変動することが知られている[7][32]。
機能
リガンド応答性
NPAS2はヘムをリガンドとして結合し、細胞内のガス状分子(CO, O2, NO)の濃度変化に応じてその立体構造や転写活性が変化する可能性が示唆されている[21][4]。これにより、NPAS2は細胞の代謝状態(例:ヘム生合成レベル)やガス環境を感知し、概日リズムや代謝関連遺伝子の発現を調節する役割を担っていると考えられる[33]。NPAS1, 3, 4も、PAS-Bドメイン内にリガンド結合ポケットを有することが構造的に示されており[16][14](図2)、これらのタンパク質は未知の内因性リガンドによって活性が制御されている可能性が考えられる。
概日リズム制御
視交叉上核(SCN)において、CLOCKと共にコア時計遺伝子(period (Per)、cryptochrome (CRY)など)の転写を制御する転写活性化因子として機能し、約24時間周期の概日リズム発振に寄与する[7][34]。末梢組織(肝臓、肺など)においても、それぞれの組織における概日時計の維持に関与する[32]。NPAS2変異マウスは、正常な光周期下では比較的正常な活動リズムを示すが、恒常暗黒下での活動周期の不安定性や、特定の光パルスに対する位相シフト反応の変化、睡眠ホメオスタシスの異常などを示す[35][36]。
代謝調節
特に肝臓において、糖新生や脂質代謝に関わる遺伝子の発現を概日的に制御し、エネルギー恒常性の維持に関与する[37][38]。
学習・記憶
NPAS2ノックアウトマウスは、文脈的恐怖条件づけや空間学習課題において記憶障害を示すことが報告されており、前脳におけるNPAS2が特定の種類の記憶形成に関与することを示唆している [39]。
疾患との関わり
睡眠障害
ヒトにおいてNPAS2遺伝子多型が睡眠相後退症候群や睡眠時間、交代勤務への耐性などに関連する可能性が報告されている[40][41]。
気分障害
概日リズムの乱れが気分障害(うつ病、双極性障害、季節性情動障害)の発症に関与することから、NPAS2遺伝子多型とこれらの疾患リスクとの関連が研究されている[42][43]。
癌
概日時計の破綻は癌リスクを増加させると考えられており、NPAS2の発現異常や遺伝子多型が乳癌、前立腺癌、大腸癌などの発症リスクや予後に関連するという報告がある[44][45][13]。NPAS2の癌における役割は、代謝制御との関連も含めて近年注目されている[38]。
NPAS3
組織分布
主に中枢神経系に限局しており、特に高レベルの発現を示す領域として、海馬(とりわけ歯状回の顆粒細胞)、嗅球、線条体、大脳皮質(特に辺縁皮質)、視床、松果体が挙げられる[30][28]。発生期の脳においてもダイナミックな発現パターンを示し、神経系の構築に重要な役割を果たすことが示唆されている[30]。
機能
神経発生
脳の発生過程において、神経幹細胞の増殖、神経細胞への分化、細胞移動、軸索伸長、および細胞生存を制御する上で極めて重要である。NPAS3ノックアウトマウスは、新生仔期に致死となる場合が多く、生存した場合でも重度の脳構造異常(特に海馬歯状回の欠損や形成不全、脳室拡大)を示し、神経発達障害のモデルとなる[30][28]。
神経伝達物質系の調節
特に中脳辺縁系ドーパミン作動性神経系の発達や機能維持に関与する可能性が、発現パターンやノックアウトマウスの表現型、精神疾患との関連から示唆されている[30]。
疾患との関わり
統合失調症・双極性障害
NPAS3は、これらの主要な精神疾患との関連が最も強く示唆されているNPASファミリーメンバーである。ヒト染色体14q13.1に位置するNPAS3遺伝子を含む領域の染色体転座が、統合失調症や重度の学習障害を持つ家系で見出されたことが最初の契機となった[9] 。その後、大規模なゲノムワイド関連解析(genome-wide association study, GWAS)や候補遺伝子関連解析により、NPAS3遺伝子内のSNPやコピー数変異(copy number variation, CNV)が統合失調症や双極性障害のリスクと有意に関連することが複数の研究で報告されている[46]。
ノックアウトマウスが示す神経発達異常や行動異常(多動性、学習障害、社会性行動の変化、抗精神病薬への反応性変化など)も、これらの疾患の病態モデルとしてNPAS3の重要性を支持している[30][28]。
知的障害・発達障害
上記の染色体異常の報告に加え、NPAS3遺伝子の変異が知的障害や発達遅延を伴う症例で報告されている[9]。実際に、脆弱X症候群(Fragile X syndrome)のモデルマウスにおいて、感覚過敏性がNPAS4依存的な抑制機能によってレスキューされることが示されている[28]。
癌
神経膠腫や肺癌など、いくつかの癌種においてNPAS3の発現変化や機能的役割が報告され始めている[13][47]。
文献[48]の図を改変)。
NPAS4
組織分布
ほぼニューロン特異的であり、特に興奮性ニューロンで顕著である[25]。成熟脳では、海馬(CA1, CA3, 歯状回)、大脳皮質の各層、扁桃体(特に基底外側核)、線条体、嗅球などで基礎レベルの発現、あるいは活動依存的な強い誘導が見られる[25][49]。神経活動、特にNMDA型グルタミン酸受容体の活性化やL型電位依存性カルシウムチャネルを通じたカルシウム流入を引き起こす刺激(例えば、新規環境探索、学習課題、薬物投与、てんかん様活動、脳虚血)によって、そのmRNAレベルが数十分から数時間以内に数十倍から数百倍にまで劇的に増加する最初期遺伝子 (immediately early gene, IEG)としての特性を持つ[50][25][51]。大脳皮質の抑制性介在ニューロンの一部(例:パルブアルブミンparvalbumin陽性細胞)や嗅球の介在ニューロンでも活動依存的な発現誘導が報告されている[52][53]。
機能
神経活動依存的な遺伝子発現のマスターレギュレーター
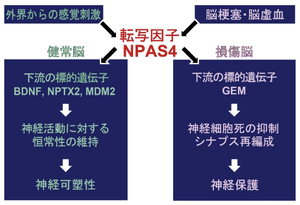
NPAS4は、興奮性ニューロンでの標的遺伝子(BDNFなど)を活性化させて、抑制性シナプスの数を増加させることで、回路全体の活動を低下させる一方、抑制性ニューロンでの標的遺伝子(MDM2など)を不活性化させて、シナプス形成を促進しGABAの放出を増加させることで、回路全体の活動を低下させるという、恒常的な可塑性を維持する役割を果たす(図3)。
ニューロンが活動すると迅速に発現が誘導され、その後、抑制性シナプスの形成や機能に関わる遺伝子群(glutamate decarboxylase 1/2 (GAD1)/2), ソマトスタチン (SST)、神経栄養因子(brain-derived neurotrophic factor, BDNF)、イオンチャネル、その他の転写因子など、多岐にわたる標的遺伝子の発現を協調的に制御する[25][49][54][52]。これにより、神経回路の活動レベルに応じた適応的な変化を引き起こす。
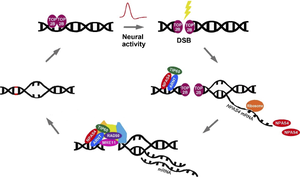
Fos、Npas4、Egr1などの最初期遺伝子(immediately early gene: IEG)のプロモーターでは、感覚刺激によりtopoisomerase IIβ(TOP2B)を介してDNA二本鎖切断(double-strand break: DSB)が形成される。マウス海馬ニューロンをカイニン酸で刺激した2時間後に観察されるDSB部位の大部分(69%)は、NPAS4/ARNTヘテロダイマーが最も多く結合しているNpas4遺伝子座と重なっていた[54](図4)。
NPAS4/ARNTとNuA4(lysine acetyltransferaseのTIP60を含む)の複合体は、神経活動により生じた二本鎖切断を、DSB修復タンパク質MRE1とRAD50をリクルートすることにより修復する(図4)。尚、Npas4プロモーターは感覚刺激により二本鎖切断を受けるが、NPAS4結合部位を含んでいるので、神経活動により誘導されたDSBをNPAS4がフィードバック制御していることになる[48]。
シナプス可塑性・学習記憶
海馬依存的な空間記憶や文脈的恐怖記憶、扁桃体依存的な手がかり恐怖記憶など、様々な種類の学習・記憶課題の遂行に必要である[55][56](。また、長期増強(long-term potentiation, LTP)や長期抑圧(long-term depression, (LTD))といったシナプス可塑性の誘導や維持にも関与することが示されている[49]。
恒常性可塑性と神経保護
興奮性ニューロンでの標的遺伝子(BDNFなど)を活性化させて、周囲の抑制性ニューロン(特にパルブアルブミン陽性細胞)への入力シナプスの数や強度を選択的に増加させる。また抑制性ニューロンでの標的遺伝子(MDM2など)を不活性化させて、シナプス形成を促進しGABAの放出を増加させる。これにより神経回路全体の興奮レベルを適切に保つ恒常性維持メカニズム(ホメオスタティックな可塑性)において中心的な役割を果たす[25][49][52][53][26](図3)。
病的条件下であるてんかん発作や脳虚血などの過剰な神経活動やストレスに応答して発現が誘導される。その結果、標的遺伝子(GEMなど)が活性化され、L型電位依存性カルシウムチャネルの細胞膜への局在をブロックすることで、細胞内へのCa2+流入を減少させ、神経細胞死を抑制するという神経保護の役割を果たす[12][57][51](図3)。
不安・恐怖応答
扁桃体におけるNPAS4の発現と機能が、不安様行動のレベルや恐怖記憶の形成・消去に関与することが示されている[56]。
疾患との関わり
てんかん
てんかん発作は強力な神経活動刺激であり、発作時にNPAS4の発現が海馬などで著しく誘導される(Lin et al., 2008)。NPAS4は抑制性シナプスの形成を促進することから、発作後の神経回路の過剰興奮を抑制する保護的な役割(抗てんかん作用)を持つと考えられている。実際に、NPAS4欠損マウスは、薬物誘発性てんかん発作に対する感受性が亢進し、発作重症度が増加することが示されている[57]。
脳梗塞・神経変性疾患
脳虚血などの神経傷害後にもNPAS4の発現が誘導され、神経保護に関与する可能性が示唆されている[12][51](図3)。その機能不全が神経変性疾患の病態に関わる可能性も研究されている。
不安障害・心的外傷後ストレス障害
扁桃体におけるNPAS4の機能は恐怖記憶の形成と消去に関与するため、その調節異常が不安障害や心的外傷後ストレス障害(posttraumatic stress disorder, PTSD)の病態に関与する可能性が考えられている[56]。
依存症
薬物(コカインなど)投与によって線条体などでNPAS4の発現が誘導され、薬物に対する報酬学習や精神刺激薬感受性、依存からの再燃に関与することが示唆されている[58]。
自閉スペクトラム症
病態仮説の一つである神経回路の興奮/抑制バランスの破綻において、NPAS4が重要な役割を果たしている可能性が注目されている。NPAS4の機能異常とASD病態との関連、および治療標的としての可能性が探求されている[59]。
関連語
- 転写因子
- Aryl hydrocarbon receptor nuclear translocator (Hypoxia-inducible factor 1β)
- Brain and muscle ARNT-like 1 (ARNT-like 1)
- Enhancer box (E-box)
- 興奮/抑制バランス
- 恒常的な可塑性
参考文献
- ↑ 1.0 1.1
Gu, Y.Z., Hogenesch, J.B., & Bradfield, C.A. (2000).
The PAS superfamily: sensors of environmental and developmental signals. Annual review of pharmacology and toxicology, 40, 519-61. [PubMed:10836146] [WorldCat] [DOI] - ↑ 2.0 2.1 2.2
Kewley, R.J., Whitelaw, M.L., & Chapman-Smith, A. (2004).
The mammalian basic helix-loop-helix/PAS family of transcriptional regulators. The international journal of biochemistry & cell biology, 36(2), 189-204. [PubMed:14643885] [WorldCat] [DOI] - ↑ 3.0 3.1
Taylor, B.L., & Zhulin, I.B. (1999).
PAS domains: internal sensors of oxygen, redox potential, and light. Microbiology and molecular biology reviews : MMBR, 63(2), 479-506. [PubMed:10357859] [PMC] [WorldCat] [DOI] - ↑ 4.0 4.1
Gilles-Gonzalez, M.A., & Gonzalez, G. (2005).
Heme-based sensors: defining characteristics, recent developments, and regulatory hypotheses. Journal of inorganic biochemistry, 99(1), 1-22. [PubMed:15598487] [WorldCat] [DOI] - ↑ 5.0 5.1 5.2 5.3
Hogenesch, J.B., Chan, W.K., Jackiw, V.H., Brown, R.C., Gu, Y.Z., Pray-Grant, M., ..., & Bradfield, C.A. (1997).
Characterization of a subset of the basic-helix-loop-helix-PAS superfamily that interacts with components of the dioxin signaling pathway. The Journal of biological chemistry, 272(13), 8581-93. [PubMed:9079689] [WorldCat] [DOI] - ↑ 6.0 6.1 6.2
Makino, Y., Kanopka, A., Wilson, W.J., Tanaka, H., & Poellinger, L. (2002).
Inhibitory PAS domain protein (IPAS) is a hypoxia-inducible splicing variant of the hypoxia-inducible factor-3alpha locus. The Journal of biological chemistry, 277(36), 32405-8. [PubMed:12119283] [WorldCat] [DOI] - ↑ 7.0 7.1 7.2 7.3 7.4
Reick, M., Garcia, J.A., Dudley, C., & McKnight, S.L. (2001).
NPAS2: an analog of clock operative in the mammalian forebrain. Science (New York, N.Y.), 293(5529), 506-9. [PubMed:11441147] [WorldCat] [DOI] - ↑
Zhou, Y.D., Barnard, M., Tian, H., Li, X., Ring, H.Z., Francke, U., ..., & McKnight, S.L. (1997).
Molecular characterization of two mammalian bHLH-PAS domain proteins selectively expressed in the central nervous system. Proceedings of the National Academy of Sciences of the United States of America, 94(2), 713-8. [PubMed:9012850] [PMC] [WorldCat] [DOI] - ↑ 9.0 9.1 9.2
Kamnasaran, D., Muir, W.J., Ferguson-Smith, M.A., & Cox, D.W. (2003).
Disruption of the neuronal PAS3 gene in a family affected with schizophrenia. Journal of medical genetics, 40(5), 325-32. [PubMed:12746393] [PMC] [WorldCat] [DOI] - ↑
Ooe, N., Saito, K., Mikami, N., Nakatuka, I., & Kaneko, H. (2004).
Identification of a novel basic helix-loop-helix-PAS factor, NXF, reveals a Sim2 competitive, positive regulatory role in dendritic-cytoskeleton modulator drebrin gene expression. Molecular and cellular biology, 24(2), 608-16. [PubMed:14701734] [PMC] [WorldCat] [DOI] - ↑
Moser, M., Knoth, R., Bode, C., & Patterson, C. (2004).
LE-PAS, a novel Arnt-dependent HLH-PAS protein, is expressed in limbic tissues and transactivates the CNS midline enhancer element. Brain research. Molecular brain research, 128(2), 141-9. [PubMed:15363889] [WorldCat] [DOI] - ↑ 12.0 12.1 12.2
Shamloo, M., Soriano, L., von Schack, D., Rickhag, M., Chin, D.J., Gonzalez-Zulueta, M., ..., & Nikolich, K. (2006).
Npas4, a novel helix-loop-helix PAS domain protein, is regulated in response to cerebral ischemia. The European journal of neuroscience, 24(10), 2705-20. [PubMed:17156197] [WorldCat] [DOI] - ↑ 13.0 13.1 13.2 13.3 13.4 13.5
Bersten, D.C., Sullivan, A.E., Peet, D.J., & Whitelaw, M.L. (2013).
bHLH-PAS proteins in cancer. Nature reviews. Cancer, 13(12), 827-41. [PubMed:24263188] [WorldCat] [DOI] - ↑ 14.0 14.1 14.2 14.3
Sun, X., Jing, L., Li, F., Zhang, M., Diao, X., Zhuang, J., ..., & Wu, D. (2022).
Structures of NPAS4-ARNT and NPAS4-ARNT2 heterodimers reveal new dimerization modalities in the bHLH-PAS transcription factor family. Proceedings of the National Academy of Sciences of the United States of America, 119(46), e2208804119. [PubMed:36343253] [PMC] [WorldCat] [DOI] - ↑
Greb-Markiewicz, B., Zarębski, M., & Ożyhar, A. (2018).
Multiple sequences orchestrate subcellular trafficking of neuronal PAS domain-containing protein 4 (NPAS4). The Journal of biological chemistry, 293(29), 11255-11270. [PubMed:29899116] [PMC] [WorldCat] [DOI] - ↑ 16.0 16.1 16.2 16.3 16.4 16.5
Wu, D., Su, X., Potluri, N., Kim, Y., & Rastinejad, F. (2016).
NPAS1-ARNT and NPAS3-ARNT crystal structures implicate the bHLH-PAS family as multi-ligand binding transcription factors. eLife, 5. [PubMed:27782878] [PMC] [WorldCat] [DOI] - ↑
Murre, C., McCaw, P.S., Vaessin, H., Caudy, M., Jan, L.Y., Jan, Y.N., ..., & Lassar, A.B. (1989).
Interactions between heterologous helix-loop-helix proteins generate complexes that bind specifically to a common DNA sequence. Cell, 58(3), 537-44. [PubMed:2503252] [WorldCat] [DOI] - ↑
Yutzey, K.E., & Konieczny, S.F. (1992).
Different E-box regulatory sequences are functionally distinct when placed within the context of the troponin I enhancer. Nucleic acids research, 20(19), 5105-13. [PubMed:1329039] [PMC] [WorldCat] [DOI] - ↑
Swanson, H.I., & Bradfield, C.A. (1993).
The AH-receptor: genetics, structure and function. Pharmacogenetics, 3(5), 213-30. [PubMed:8287061] [WorldCat] [DOI] - ↑
Hirose, K., Morita, M., Ema, M., Mimura, J., Hamada, H., Fujii, H., ..., & Fujii-Kuriyama, Y. (1996).
cDNA cloning and tissue-specific expression of a novel basic helix-loop-helix/PAS factor (Arnt2) with close sequence similarity to the aryl hydrocarbon receptor nuclear translocator (Arnt). Molecular and cellular biology, 16(4), 1706-13. [PubMed:8657146] [PMC] [WorldCat] [DOI] - ↑ 21.0 21.1
Dioum, E.M., Rutter, J., Tuckerman, J.R., Gonzalez, G., Gilles-Gonzalez, M.A., & McKnight, S.L. (2002).
NPAS2: a gas-responsive transcription factor. Science (New York, N.Y.), 298(5602), 2385-7. [PubMed:12446832] [WorldCat] [DOI] - ↑
Ascenzi, P., Bocedi, A., Leoni, L., Visca, P., Zennaro, E., Milani, M., & Bolognesi, M. (2004).
CO sniffing through heme-based sensor proteins. IUBMB life, 56(6), 309-15. [PubMed:15370879] [WorldCat] [DOI] - ↑ 23.0 23.1
Luoma, L.M., & Berry, F.B. (2018).
Molecular analysis of NPAS3 functional domains and variants. BMC molecular biology, 19(1), 14. [PubMed:30509165] [PMC] [WorldCat] [DOI] - ↑
Kondratov, R.V., Chernov, M.V., Kondratova, A.A., Gorbacheva, V.Y., Gudkov, A.V., & Antoch, M.P. (2003).
BMAL1-dependent circadian oscillation of nuclear CLOCK: posttranslational events induced by dimerization of transcriptional activators of the mammalian clock system. Genes & development, 17(15), 1921-32. [PubMed:12897057] [PMC] [WorldCat] [DOI] - ↑ 25.0 25.1 25.2 25.3 25.4 25.5
Lin, Y., Bloodgood, B.L., Hauser, J.L., Lapan, A.D., Koon, A.C., Kim, T.K., ..., & Greenberg, M.E. (2008).
Activity-dependent regulation of inhibitory synapse development by Npas4. Nature, 455(7217), 1198-204. [PubMed:18815592] [PMC] [WorldCat] [DOI] - ↑ 26.0 26.1
Sun, X., & Lin, Y. (2016).
Npas4: Linking Neuronal Activity to Memory. Trends in neurosciences, 39(4), 264-275. [PubMed:26987258] [PMC] [WorldCat] [DOI] - ↑
Eckel-Mahan, K., & Sassone-Corsi, P. (2013).
Metabolism and the circadian clock converge. Physiological reviews, 93(1), 107-35. [PubMed:23303907] [PMC] [WorldCat] [DOI] - ↑ 28.0 28.1 28.2 28.3 28.4 28.5
Michaelson, J.J., Shin, M.K., Koh, J.Y., Brueggeman, L., Zhang, A., Katzman, A., ..., & Pieper, A.A. (2017).
Neuronal PAS Domain Proteins 1 and 3 Are Master Regulators of Neuropsychiatric Risk Genes. Biological psychiatry, 82(3), 213-223. [PubMed:28499489] [PMC] [WorldCat] [DOI] - ↑
Lopez-Mejia, A., Moreno-Londoño, A.P., Fonseca Camarillo, G., Yamamoto-Furusho, J.K., Villanueva-Herrero, J.A., de Leon-Rendón, J.L., ..., & Robles-Flores, M. (2025).
Hypoxia inducible factor 3-alpha promotes a malignant phenotype in colorectal cancer cells. IUBMB life, 77(2), e70007. [PubMed:39981666] [PMC] [WorldCat] [DOI] - ↑ 30.0 30.1 30.2 30.3 30.4 30.5
Brunskill, E.W., Ehrman, L.A., Williams, M.T., Klanke, J., Hammer, D., Schaefer, T.L., ..., & Vorhees, C.V. (2005).
Abnormal neurodevelopment, neurosignaling and behaviour in Npas3-deficient mice. The European journal of neuroscience, 22(6), 1265-76. [PubMed:16190882] [WorldCat] [DOI] - ↑
Levesque, B.M., Zhou, S., Shan, L., Johnston, P., Kong, Y., Degan, S., & Sunday, M.E. (2007).
NPAS1 regulates branching morphogenesis in embryonic lung. American journal of respiratory cell and molecular biology, 36(4), 427-34. [PubMed:17110583] [PMC] [WorldCat] [DOI] - ↑ 32.0 32.1 32.2
Storch, K.F., Lipan, O., Leykin, I., Viswanathan, N., Davis, F.C., Wong, W.H., & Weitz, C.J. (2002).
Extensive and divergent circadian gene expression in liver and heart. Nature, 417(6884), 78-83. [PubMed:11967526] [WorldCat] [DOI] - ↑
Kitanishi, K., Igarashi, J., Hayasaka, K., Hikage, N., Saiful, I., Yamauchi, S., ..., & Shimizu, T. (2008).
Heme-binding characteristics of the isolated PAS-A domain of mouse Per2, a transcriptional regulatory factor associated with circadian rhythms. Biochemistry, 47(23), 6157-68. [PubMed:18479150] [WorldCat] [DOI] - ↑
Parekh, P.K., Logan, R.W., Ketchesin, K.D., Becker-Krail, D., Shelton, M.A., Hildebrand, M.A., ..., & McClung, C.A. (2019).
Cell-Type-Specific Regulation of Nucleus Accumbens Synaptic Plasticity and Cocaine Reward Sensitivity by the Circadian Protein, NPAS2. The Journal of neuroscience : the official journal of the Society for Neuroscience, 39(24), 4657-4667. [PubMed:30962277] [PMC] [WorldCat] [DOI] - ↑
Dudley, C.A., Erbel-Sieler, C., Estill, S.J., Reick, M., Franken, P., Pitts, S., & McKnight, S.L. (2003).
Altered patterns of sleep and behavioral adaptability in NPAS2-deficient mice. Science (New York, N.Y.), 301(5631), 379-83. [PubMed:12843397] [WorldCat] [DOI] - ↑
Mongrain, V., La Spada, F., Curie, T., & Franken, P. (2011).
Sleep loss reduces the DNA-binding of BMAL1, CLOCK, and NPAS2 to specific clock genes in the mouse cerebral cortex. PloS one, 6(10), e26622. [PubMed:22039518] [PMC] [WorldCat] [DOI] - ↑
Lee, S.M., Zhang, Y., Tsuchiya, H., Smalling, R., Jetten, A.M., & Wang, L. (2015).
Small heterodimer partner/neuronal PAS domain protein 2 axis regulates the oscillation of liver lipid metabolism. Hepatology (Baltimore, Md.), 61(2), 497-505. [PubMed:25212631] [PMC] [WorldCat] [DOI] - ↑ 38.0 38.1
Ma, S., Chen, Y., Quan, P., Zhang, J., Han, S., Wang, G., ..., & Qin, W. (2023).
NPAS2 promotes aerobic glycolysis and tumor growth in prostate cancer through HIF-1A signaling. BMC cancer, 23(1), 280. [PubMed:36978001] [PMC] [WorldCat] [DOI] - ↑
Garcia, J.A., Zhang, D., Estill, S.J., Michnoff, C., Rutter, J., Reick, M., ..., & McKnight, S.L. (2000).
Impaired cued and contextual memory in NPAS2-deficient mice. Science (New York, N.Y.), 288(5474), 2226-30. [PubMed:10864874] [WorldCat] [DOI] - ↑
Evans, D.S., Parimi, N., Nievergelt, C.M., Blackwell, T., Redline, S., Ancoli-Israel, S., ..., & Osteoporotic Fractures in Men (MrOS) Study Group (2013).
Common genetic variants in ARNTL and NPAS2 and at chromosome 12p13 are associated with objectively measured sleep traits in the elderly. Sleep, 36(3), 431-46. [PubMed:23449886] [PMC] [WorldCat] [DOI] - ↑
Dall'Ara, I., Ghirotto, S., Ingusci, S., Bagarolo, G., Bertolucci, C., & Barbujani, G. (2016).
Demographic history and adaptation account for clock gene diversity in humans. Heredity, 117(3), 165-72. [PubMed:27301334] [PMC] [WorldCat] [DOI] - ↑
Mansour, H.A., Wood, J., Logue, T., Chowdari, K.V., Dayal, M., Kupfer, D.J., ..., & Nimgaonkar, V.L. (2006).
Association study of eight circadian genes with bipolar I disorder, schizoaffective disorder and schizophrenia. Genes, brain, and behavior, 5(2), 150-7. [PubMed:16507006] [WorldCat] [DOI] - ↑
Soria, V., Martínez-Amorós, E., Escaramís, G., Valero, J., Pérez-Egea, R., García, C., ..., & Urretavizcaya, M. (2010).
Differential association of circadian genes with mood disorders: CRY1 and NPAS2 are associated with unipolar major depression and CLOCK and VIP with bipolar disorder. Neuropsychopharmacology : official publication of the American College of Neuropsychopharmacology, 35(6), 1279-89. [PubMed:20072116] [PMC] [WorldCat] [DOI] - ↑
Zhu, Y., Stevens, R.G., Leaderer, D., Hoffman, A., Holford, T., Zhang, Y., ..., & Zheng, T. (2008).
Non-synonymous polymorphisms in the circadian gene NPAS2 and breast cancer risk. Breast cancer research and treatment, 107(3), 421-5. [PubMed:17453337] [PMC] [WorldCat] [DOI] - ↑
Yi, C., Mu, L., de la Longrais, I.A., Sochirca, O., Arisio, R., Yu, H., ..., & Katsaro, D. (2010).
The circadian gene NPAS2 is a novel prognostic biomarker for breast cancer. Breast cancer research and treatment, 120(3), 663-9. [PubMed:19649706] [PMC] [WorldCat] [DOI] - ↑
Pickard, B.S., Christoforou, A., Thomson, P.A., Fawkes, A., Evans, K.L., Morris, S.W., ..., & Muir, W.J. (2009).
Interacting haplotypes at the NPAS3 locus alter risk of schizophrenia and bipolar disorder. Molecular psychiatry, 14(9), 874-84. [PubMed:18317462] [WorldCat] [DOI] - ↑
Yu, B., Xing, Z., Tian, X., & Feng, R. (2024).
A Prognostic Risk Signature of Two Autophagy-Related Genes for Predicting Triple-Negative Breast Cancer Outcomes. Breast cancer (Dove Medical Press), 16, 529-544. [PubMed:39246674] [PMC] [WorldCat] [DOI] - ↑ 48.0 48.1
Delint-Ramirez, I., & Madabhushi, R. (2023).
NPAS4 juggles neuronal activity-dependent transcription and DSB repair with NuA4. Molecular cell, 83(8), 1208-1209. [PubMed:37084713] [WorldCat] [DOI] - ↑ 49.0 49.1 49.2 49.3
Bloodgood, B.L., Sharma, N., Browne, H.A., Trepman, A.Z., & Greenberg, M.E. (2013).
The activity-dependent transcription factor NPAS4 regulates domain-specific inhibition. Nature, 503(7474), 121-5. [PubMed:24201284] [PMC] [WorldCat] [DOI] - ↑
Flavell, S.W., Cowan, C.W., Kim, T.K., Greer, P.L., Lin, Y., Paradis, S., ..., & Greenberg, M.E. (2006).
Activity-dependent regulation of MEF2 transcription factors suppresses excitatory synapse number. Science (New York, N.Y.), 311(5763), 1008-12. [PubMed:16484497] [WorldCat] [DOI] - ↑ 51.0 51.1 51.2
Takahashi, H., Asahina, R., Fujioka, M., Matsui, T.K., Kato, S., Mori, E., ..., & Tsuboi, A. (2021).
Ras-like Gem GTPase induced by Npas4 promotes activity-dependent neuronal tolerance for ischemic stroke. Proceedings of the National Academy of Sciences of the United States of America, 118(32). [PubMed:34349016] [PMC] [WorldCat] [DOI] - ↑ 52.0 52.1 52.2
Spiegel, I., Mardinly, A.R., Gabel, H.W., Bazinet, J.E., Couch, C.H., Tzeng, C.P., ..., & Greenberg, M.E. (2014).
Npas4 regulates excitatory-inhibitory balance within neural circuits through cell-type-specific gene programs. Cell, 157(5), 1216-29. [PubMed:24855953] [PMC] [WorldCat] [DOI] - ↑ 53.0 53.1
Yoshihara, S., Takahashi, H., Nishimura, N., Kinoshita, M., Asahina, R., Kitsuki, M., ..., & Tsuboi, A. (2014).
Npas4 regulates Mdm2 and thus Dcx in experience-dependent dendritic spine development of newborn olfactory bulb interneurons. Cell reports, 8(3), 843-57. [PubMed:25088421] [WorldCat] [DOI] - ↑ 54.0 54.1
Pollina, E.A., Gilliam, D.T., Landau, A.T., Lin, C., Pajarillo, N., Davis, C.P., ..., & Greenberg, M.E. (2023).
A NPAS4-NuA4 complex couples synaptic activity to DNA repair. Nature, 614(7949), 732-741. [PubMed:36792830] [PMC] [WorldCat] [DOI] - ↑
Ramamoorthi, K., Fropf, R., Belfort, G.M., Fitzmaurice, H.L., McKinney, R.M., Neve, R.L., ..., & Lin, Y. (2011).
Npas4 regulates a transcriptional program in CA3 required for contextual memory formation. Science (New York, N.Y.), 334(6063), 1669-75. [PubMed:22194569] [PMC] [WorldCat] [DOI] - ↑ 56.0 56.1 56.2
Ploski, J.E., Monsey, M.S., Nguyen, T., DiLeone, R.J., & Schafe, G.E. (2011).
The neuronal PAS domain protein 4 (Npas4) is required for new and reactivated fear memories. PloS one, 6(8), e23760. [PubMed:21887312] [PMC] [WorldCat] [DOI] - ↑ 57.0 57.1
Shan, W., Nagai, T., Tanaka, M., Itoh, N., Furukawa-Hibi, Y., Nabeshima, T., ..., & Yamada, K. (2018).
Neuronal PAS domain protein 4 (Npas4) controls neuronal homeostasis in pentylenetetrazole-induced epilepsy through the induction of Homer1a. Journal of neurochemistry, 145(1), 19-33. [PubMed:29222951] [WorldCat] [DOI] - ↑
Taniguchi, M., Carreira, M.B., Cooper, Y.A., Bobadilla, A.C., Heinsbroek, J.A., Koike, N., ..., & Cowan, C.W. (2017).
HDAC5 and Its Target Gene, Npas4, Function in the Nucleus Accumbens to Regulate Cocaine-Conditioned Behaviors. Neuron, 96(1), 130-144.e6. [PubMed:28957664] [PMC] [WorldCat] [DOI] - ↑
Rein, B., Tan, T., Yang, F., Wang, W., Williams, J., Zhang, F., ..., & Yan, Z. (2021).
Reversal of synaptic and behavioral deficits in a 16p11.2 duplication mouse model via restoration of the GABA synapse regulator Npas4. Molecular psychiatry, 26(6), 1967-1979. [PubMed:32099100] [PMC] [WorldCat] [DOI]